ChIP-Seq Analysis Tutorial
ChIP-Seq の簡単な紹介
タンパク質と DNA の相互作用は、細胞生理学の根底にあるメカニズムを解明するために広く使用されています。 クロマチン免疫沈降 (ChIP) アッセイ技術の開発により、このようなメカニズムの研究が可能になりました。 さらなる開発の後、特異性と感度の点で利点を提供するディープシーケンシングの代替法 (ChiP-Seq) が登場しました。
ChIP-Seq 実験は、ホルムアルデヒドによる細胞全体の架橋から始まり、その後超音波処理と DNA 単離が続きます。 その後、特定のタンパク質に結合する抗体からなる DNA-タンパク質複合体の免疫沈降が実行されます。 形成された免疫複合体は沈殿され、精製されます。 最後に、DNA の配列が決定され、濃縮された部位の高解像度データが生成されます。 このアプローチと十分に確立された ChIP-seq パイプラインを組み合わせることで、研究者は DNA 転写因子、ヒストン修飾部位、エピジェネティックな変化、および遺伝子制御ネットワークのサインを捕捉することができます。
臨床的関連性と応用
疾患や健康状態にわたるエピジェネティックな不均衡には、ヒストン修飾や転写因子の変化が関与している可能性があります。 ここでは、ChIP-Seq 研究は、癌やその他の疾患の根底にある病理学的分子機構を解明するために使用されています。 ChIP-seq 解析は、疾患時の転写因子の役割の理解にも貢献します。 実際、一部の転写物は臨床表現型の発現中に変化するようです。
ChIP-Seq パイプラインの概要
ChIP-Seq 解析パイプラインは DNA-タンパク質相互作用プロジェクトの主要コンポーネントであり、生データ処理、品質管理解析、参照ゲノムとのアライメント、アライメントされたリードの品質チェック、ピーク コール、アノテーション、および視覚化。 ただし、ChIP-seq 実験で高品質の結果を得るには、考え抜かれた実験デザインを行うことが重要です。 分析を開始する前に、サンプルの複製、コントロール グループ、シーケンス キット、シーケンス プラットフォームなどのパラメーターを考慮することが重要です。
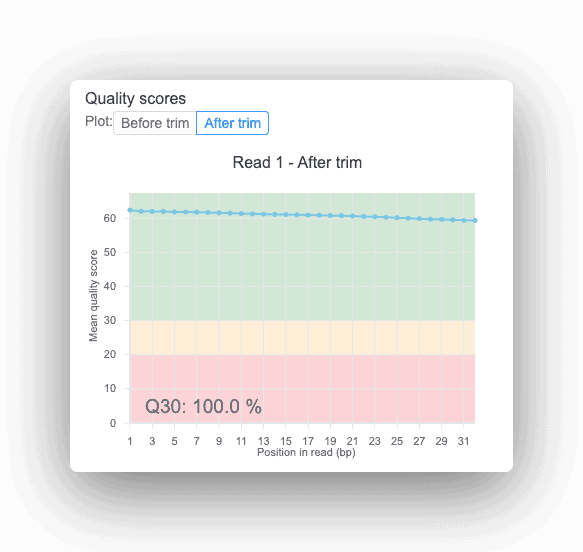
品質管理
すべての Basepair レポートは、潜在的なシーケンスの問題や入力データの汚染を明らかにするのに役立つ品質スコアを提供します。
品質管理 (QC) ステップは、シーケンスから生成されたハイスループット データの品質を評価することを目的としています。 このステップは、DNA-seq および RNA-seq 解析で実行されるステップと同様です。 ここで評価される主な指標には、配列と塩基の品質、GC 含有量、配列決定アダプターの存在、および過剰表現された配列が含まれます。 このタイプの分析で最も一般的に使用されるプログラムの 1 つは FastQC です。 さらに、低品質のシーケンスが特定された場合は、後でトリミングステップ中にそれらを削除することができます。 これはオプションの手順ですが、トリミングにより高品質の読み取りのみが保持されるため、データ品質が向上します。
位置合わせ
QC 測定後、ChIP-Seq リードは参照ゲノムとアライメントされます。 リードマッピングにより、研究者はゲノム内のリード配列の起源を特定できます。 使用される一般的なアライメント ソフトウェア ツールには、Bowtie と BWA があり、どちらも Basepair の ChIP-seq パイプラインで使用されます。 どちらのツールも、参照ゲノムに対して発散性の低い配列をマッピングします。
読み取りカウントのフローは、トリミング、アラインメント、および重複排除プロセスの最後に使用可能な読み取りの全体像を提供するのに役立ちます。 この図をデータ分析の組み立てラインとして考えてください。生データを入力し、使用可能な読み取りの出力を取得します。
アラインメントされたリードの品質チェック
次のステップは、位置合わせされたデータセットの QC 推論で構成されます。 マッピングプロセス中に、PCR 増幅およびシーケンスによって導入されたリード重複により、ピークコールおよびエンリッチメント分析中にバイアスが発生します。 Basepair は、Picard ツールを使用して重複を削除します。 重複を削除したら、アライメントされたリードの非冗長部分 (NRF) を評価する必要があります。 NRF は、参照ゲノムにマッピングされる固有のリードを測定します。 理想的な ChIP-seq 実験では、ポジションごとにリードが 3 つ未満である必要があります。
ピークコーリング
ピークコーリングステップでは、ゲノム上の濃縮されたタンパク質-DNA 相互作用領域を検出します。 Basepair の ChIP-seq パイプラインは、MACS2 を使用してこの分析を実行します。 MACS2 では、ピーク コールは 3 つの主要なステップに基づいて実行されます。フラグメント推定、それに続くローカル ノイズ パラメータの特定、そしてピークの特定です。 このステップの出力として、ユーザーは、エンリッチメント スコア、-log10pvalue、-log10qvalue、ピーク開始位置などのピーク情報を含む最終テーブルを取得します。 このステップでは、調査対象のデータセットと比較するために、コントロール サンプルの使用を強くお勧めします。 優れた対照グループはより信頼性の高い結果をもたらすことに留意してください。
各ピークにはプロモーター、イントロン、または遺伝子間の注釈が付けられ、対応する遺伝子が表示されます。 見つかったピークについては、過剰に存在する転写因子結合部位を見つけるためにモチーフ解析が行われます。
結果の概要
ChIP-seq パイプラインは、クロマチンの状態に関する情報だけでなく、決定された遺伝子または遺伝子座のコンテキストにおける転写因子の結合に関する情報も提供できます。 DNA 調節領域におけるヒストン修飾と転写因子の発生は、状態特異的なエピジェネティックな特徴を構成する可能性があります。 したがって、エピジェネティックな摂動は臨床表現型に関連している可能性があります。 たとえば、クロマチン状態の不均一性は、乳がんの治療抵抗性につながる可能性があります。 これらの細胞は抑制性ヒストン修飾マーカーを失い、がん治療に対する耐性を促進することが知られている遺伝子の発現をさらに増加させる傾向があります。
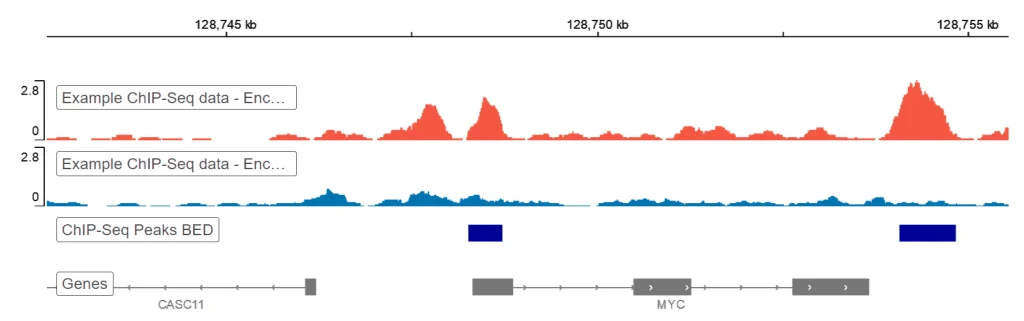
ChIP-Seq 解析パイプラインにおけるピーク、モチーフ、パスウェイ解析
モチーフ転写因子濃縮の同定は、転写因子が特定の領域で協調しているか競合しているかを解明するために使用されます。 DNA モチーフ領域のピークを同定すると、実験結果の解釈が改善されます。 ピーク分析とモチーフ分析の両方を組み合わせることで、細胞内で何が起こっているのかについての洞察が得られます。 ピークとモチーフの濃縮を統合すると、生物学的影響の可能性があるエピゲノムのランドスケープが得られます。 さらに、経路解析は、経路内のタンパク質を同定するために使用されます。 調査と結論はタンパク質の存在に基づいて定式化されます。
データの視覚化
ChIP-seq パイプラインから得られたデータは、ゲノム ブラウザーを使用して視覚化できます。 Basepair レポートには、データを操作できる組み込み IGV2 ゲノム ブラウザーが含まれています。 データは、特定のマークの有無を示すデータ密度に基づく代表的な強度インフォグラフィックであるヒートマップを使用して視覚化することもできます。 ここで使用される他のグラフィックスには、エンリッチメント プロット、upSet、およびゲノム上のピーク領域のカバレッジを計算して表示するカバレッジ プロットが含まれます。
ゲノム ブラウザは、生のゲノム データを視覚化するための優れたツールです。 これは、Basepair のすべての ChIP-seq 分析レポートに組み込まれています。
References
1. Grosselin, K., A. Durand, et al. High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer. Nat Genet, v.51, n.6, Jun, p.1060-1066. 2019.
2. Northrup, D. L. e K. Zhao. Application of ChIP-Seq and related techniques to the study of immune function. Immunity, v.34, n.6, Jun 24, p.830-42. 2011.
3. Park, S. J., J. H. Kim, et al. A ChIP-Seq Data Analysis Pipeline Based on Bioconductor Packages. Genomics Inform, v.15, n.1, Mar, p.11-18. 2017.
4. Pepke, S., B. Wold, et al. Computation for ChIP-seq and RNA-seq studies. Nat Methods, v.6, n.11 Suppl, Nov, p.S22-32. 2009.
5. Satoh, J., N. Kawana, et al. Pathway Analysis of ChIP-Seq-Based NRF1 Target Genes Suggests a Logical Hypothesis of their Involvement in the Pathogenesis of Neurodegenerative Diseases. Gene Regul Syst Bio, v.7, p.139-52. 2013.
