Error ID 11 – Failed to estimate expression variance
エラーの説明
差分式パイプラインを実行しようとしたが、サンプル内の式のバリエーションが少なすぎるため、このエラーが発生しました。 これにはいくつかの潜在的な原因が考えられます。(1) 遺伝子にマッピングされているリード数が少なすぎるため、発現推定の信頼性が低すぎる、または (2) サンプル間の実際の生物学的差異は非常にわずかであるか、です。
解決策
解決策 1
サンプルを適切なグループに割り当てたことを再確認してください。
解決策 2
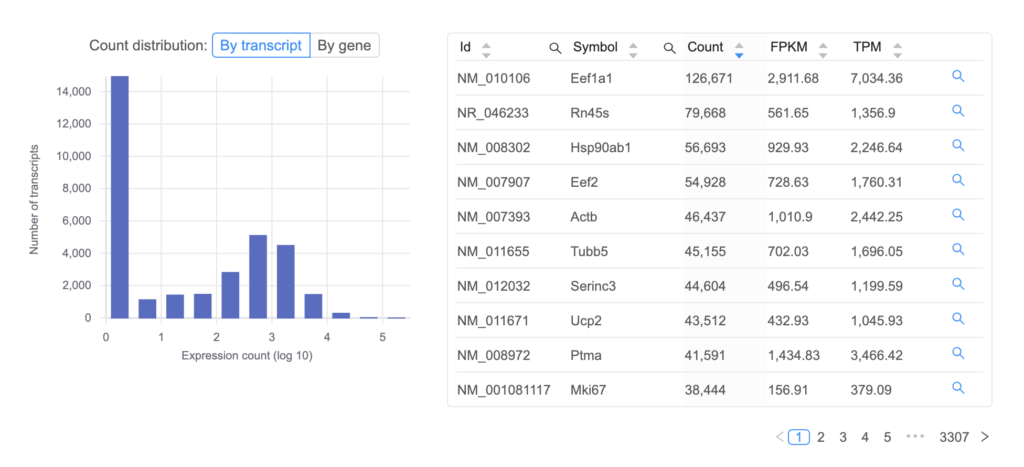
リードを調整して遺伝子発現を推定したパイプライン (「Expression count (STAR)」パイプラインなど) を再確認します。 分析ページの下部に、遺伝子ごとの発現推定値の表が表示されます (画像例を参照)。 [Count] 列を確認すると、各遺伝子にマッピングされたリードの数が表示されます。 たとえば、大多数の遺伝子のカウントが 10 以下である場合は、信頼できる発現推定値を取得するのに、このサンプルには十分なリード数がない可能性があります。 この場合、いくつかのオプションがあります。
- より高いシーケンス深度でサンプルを再シーケンスしなおす。
- パイプラインを再実行し、式の推定にマルチマップされた読み取りを含めるようにします (ただし、これはお勧めできません)。
- トリミングステップでリードの多くが削除されている場合は、サンプルの品質が低いことを示している可能性があります。 ただし、発現カウントパイプラインを再実行して、トリミングしきい値パラメーターの厳密性をわずかに下げることができます。 ただし、これもあまりお勧めできません。
解決策 3 – お問い合わせください
それでも問題が解決できない場合は、次のいずれかの方法でお気軽にお問い合わせください。
- ここでチケットを作成します: http://support.basepairtech.com/support/tickets/new
- Basepair ダッシュボードの画面右下隅にあるチャット アイコンを使用してメッセージを送信してください。
さらに詳しい情報
「発現差解析(DESeq、2グループの比較)」パイプラインまたは「発現差 (DESeq、3+ グループの比較)」パイプラインのいずれかを実行した場合、基礎となるソフトウェア (DESeq2) は、分散推定を提供するためのさらにいくつかの代替手段を提供します。 ただし、Basepairは現在、これらの他のオプションをサポートしていませんが、こちらで説明されています:https://bioconductor.org/packages/release/bioc/html/DESeq2.html
