Error ID 2 – No peaks found
エラーの説明
このエラーは、ピーク呼び出しパイプラインの 1 つを使用したものの、ピークが見つからなかった場合に発生します。
これにはさまざまな原因と解決策が考えられます。 多くの場合、これはそれほど厳密ではないパラメーターを使用することで解決できますが、ここでのリスクは、識別されたピークが偽陽性である可能性が高いことです。 もう1つの潜在的な原因 (コントロールを使用している場合)は、テストとコントロールの差がが十分でない (そのため、ピークが見つからない))ことです。 たとえば、十分な抗体が使用されなかった場合、または抗体があまり良好ではなかった場合に、これが発生する可能性があります。 最悪のシナリオは、より多くの実験を行い、より多くのデータをシーケンスする必要があることです。
解決策
問題を解決するか、少なくとも問題をより深く理解するのに役立つさまざまな解決策を次に示します。
解決策 1 – より緩やかなパラメータ
パイプラインは MACSソフトウェアを使用しており、変更できるパラメーターがいくつかあります。 すべては、分析設定ページの「その他のオプション」ボタンにリストされています。
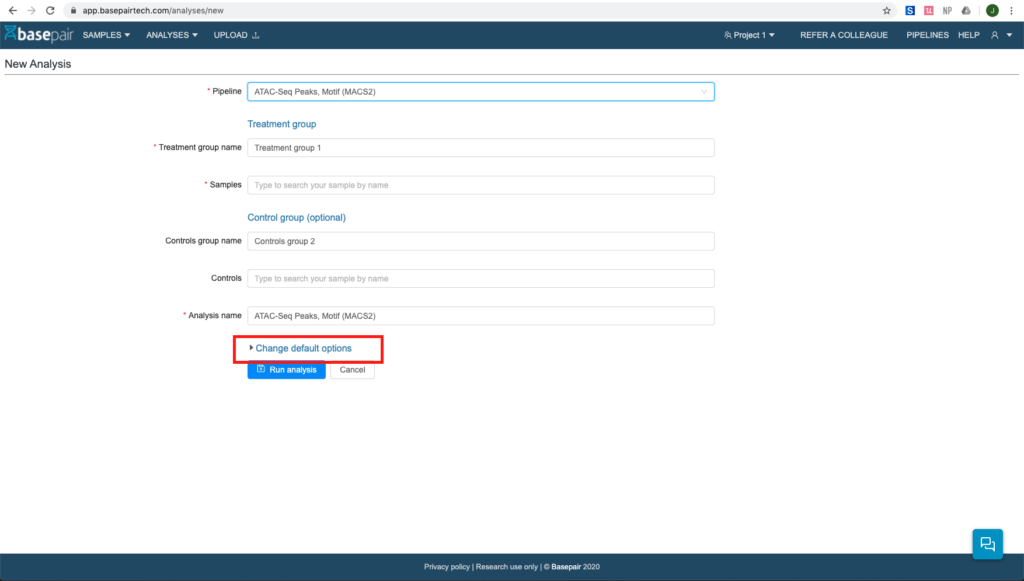
次のことを試すことができます。
- P値カットオフ : これを 1e-4 や 1e-3 などのより高い値に変更できます。
- MFOLD (低め) : 値5が推奨されますが、これを 4、3、または 2 に下げることもできます。
解決策 2 – ブラックリストフィルタリングから何が削除されたかを確認する (ChIP-seqのみ)
ChIP-seqピーク コールで実行される一般的な手順は、ブラックリストフィルタリングです。 ブラックリストは、偽陽性ピークが一般的に含まれると特定された一連の領域です (詳細については、こちら https://www.ncbi.nlm.nih.gov/pubmed/31249361 を参照してください)。 したがって、ブラックリスト領域と重なるピークは削除されます。
確認できることの 1 つは、ブラックリストフィルタリングの前にピークが呼び出されたかどうかを確認することです。 「macs2_peaks.xls」で終わるファイルを検索すると、呼び出された生のピークのセットを表示できます。 そこにピークが見られる場合は、ブラックリストフィルタリングによってすべてが削除されたことを意味します。 したがって、これはサンプルの品質が低いことを示している可能性があり、(1) 上記の解決策1の提案を試すか、(2) 一部のピークが強く思われる場合は、オプションでブラックリストフィルタリングをオフにすることができます (非推奨)。それらの領域は本物です。 (2) については、分析を設定するときに「デフォルトオプションの変更」でデフォルト パラメータを変更することでこれを行うことができます。 「 ブラックリストフィルタリングを有効にする」をFalseに設定します。
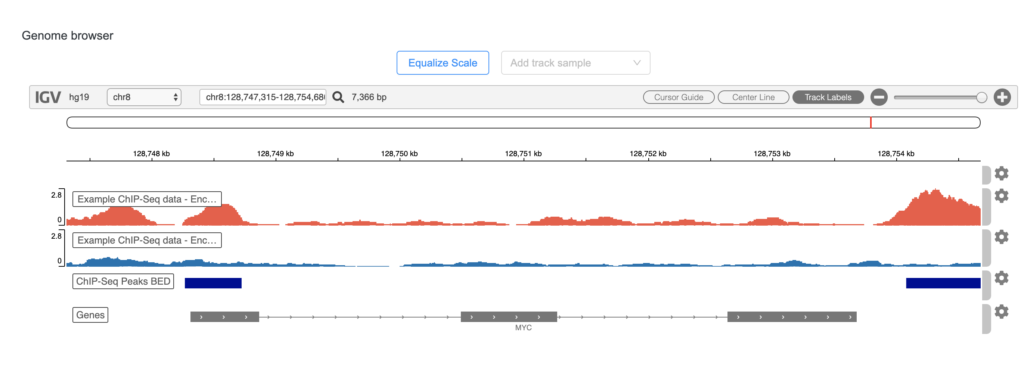
解決策 3 – IGV カバレッジ トラックを比較する (コントロールを使用している場合)
インプットコントロールを使用している場合は、「ゲノムブラウザ」を使用してカバレッジ トラックを比較します。 これを行うと、テストサンプルと入力コントロールサンプルが世界規模でどの程度「類似している」かを明らかにすることができます。 それらが (カバレッジの場所と信号強度の点で) 非常に似ている場合、ワークフローはピークを検出できない可能性があります。 この原因は、データの生成方法に応じてさまざまである可能性があります (ChIP-seq で十分な抗体が使用されなかったなど)。 解決策1で提案されているように、使用する文字列パラメータを減らしてみることができます。
解決策 4 – お問い合わせください
それでも問題が解決できない場合は、次のいずれかの方法でお気軽にお問い合わせください。
- ここでチケットを作成します: http://support.basepairtech.com/support/tickets/new
- Basepair ダッシュボードの画面右下隅にあるチャット アイコンを使用してメッセージを送信してください。
