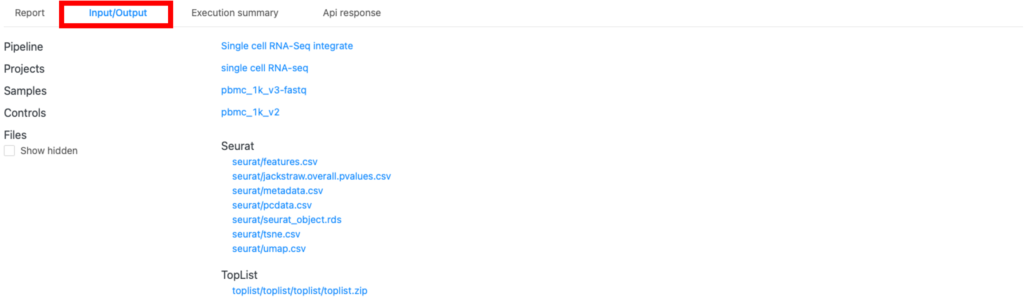
IntegrateパイプラインでSeuratが生成するアウトプットファイルについて説明します。アウトプットファイルは、「Input/output」タブ(下図の赤枠)にあります。
Seurat
| ファイル名 | ファイルの説明 |
| seurat/features.csv | Seurat 解析で同定されたフィーチャー/遺伝子のリスト(カンマ区切り形式) |
| seurat/jackstraw.overall.pvalues.csv | SeuratのJackStraw分析によって生成された各Principal component(PC)のスコア。このスコアは、各PCの有意性を決定するために使用されます(カンマ区切り形式) |
| seurat/metadata.csv | 処理後のSeuratオブジェクトの各細胞のメタデータ。処理にはフィルタリング、正規化、クラスタリングが含まれます。細胞あたりのカウント数や遺伝子・特徴などの情報が含まれます(カンマ区切り形式) |
| seurat/pcdata.csv | 各細胞のPCスコア。これらはSeuratのPCAステップから得られたものです(カンマ区切り形式) |
Seurat
| ファイル名 | ファイルの説明 |
| seurat/seurat_object.rds | RDS(R Data Serialization)ファイル。処理・解析後のSeuratオブジェクト全体を保存しています。生データと正規化されたデータとメタデータが含まれています |
| seurat/tsne.csv | 各細胞のt-SNE座標。t-SNEは非線形次元削減法です。2D・3D空間における細胞のクラスターを可視化するために使用されます(カンマ区切り形式) |
| seurat/umap.csv | 各細胞の UMAP 座標。UMAPも細胞のクラスターを可視化する技術です(カンマ区切り形式) |
TopList
| ファイル名 | ファイルの説明 |
| toplist/toplist/toplist/toplist.zip | 同定されたマーカー遺伝子を含むzipファイル。0.1-2.0の解像度のヒートマップも提供します |
Seuratセクションのアウトプットファイル
seurat_features.csv
このファイルには、Seurat解析で同定されたフィーチャー・遺伝子のリストが含まれています(カンマ区切り形式)。

用語
| 用語 | 定義 |
| Gene | 特定のデータベースで特定の遺伝子に割り当てられたユニークな識別子。例えば、この場合のEnsemblデータ |
| Gene_name | 遺伝子記号 |
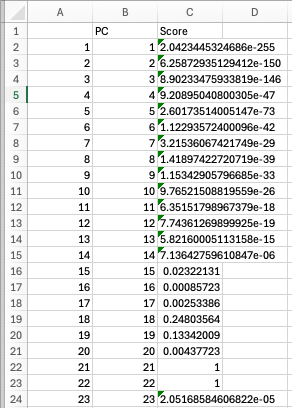
seurat_jackstraw.overall.pvalues.csv
このファイルは、SeuratのJackStraw解析中に生成されます。ファイルはカンマ区切り形式です。
JackStraw法は、Principal Component Analysis(PCA)のprincipal component(PC)の有意性を評価するために使用されます。この結果は、どのPCを下流のクラスタリングステップに含めるべきかを決定するのに役立ちます。
用語
| 用語 | 定義 |
| PC | PCAから得られたprincipal component(PC)数 |
| Score | 各PCのJackStrawスコア。このスコアは、PCがデータセット全体の変動にどれだけ強く寄与しているかを表しています。また、各PCの有意性も示しています |
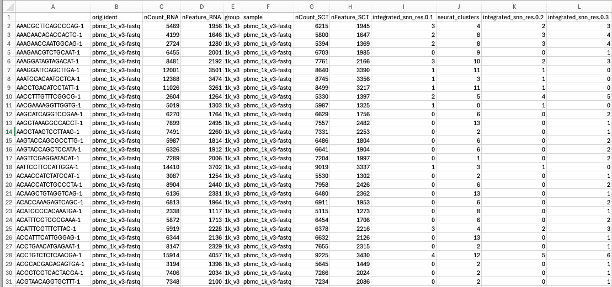
seurat/metadata.csv
このファイルには、処理後のSeuratオブジェクトの各セルのメタデータが含まれます。処理にはフィルタリング、正規化、クラスタリングが含まれます。ファイルはカンマ区切り形式です。
用語
| 用語 | 定義 |
| Orig.ident | 各細胞のサンプル名 |
| nCount_RNA | 細胞あたりのカウント数 |
| nFeature_RNA | 各細胞で検出された遺伝子・特徴の数 |
| Group_source | サンプルグループ名 |
| nCount_SCT | SCTransform正規化法によって正規化されたカウント |
| nFeature_SCT | SCTransform正規化法で正規化された遺伝子・特徴数 |
| Integrated_snn_res.x | 各細胞が特定の解像度で割り当てられたクラスター。この割り当てはShared Nearest Neighbor (SNN)クラスタリングによって行われます。「x」は、クラスタリングで使用される解像度パラメータを表します |
| Seurat_clusters | クラスタリング後に各細胞が割り当てられたクラスター。これはSeuratによる最適解像度に基づいたデフォルトの割り当てです |
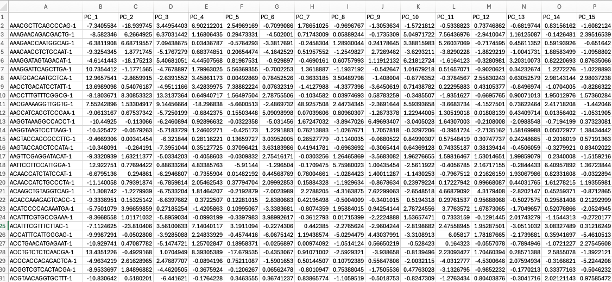
seurat_pcdata.csv
このファイルには各セルのPCスコアが含まれています。これらはSeuratのPCAステップから得られたものです。ファイルはカンマ区切り形式です。
用語
| 用語 | 定義 |
| PC_x | PCAによって生成された各PCのPCスコア。スコアの大きさは、特定の細胞の遺伝子発現プロファイルが特定のPCにどれだけ寄与しているかを反映します正のスコア:細胞の遺伝子発現プロファイルが、特定のPCによって提示された変動と強く一致していることを示唆します負のスコア:その細胞の遺伝子発現プロファイルが逆の意味で明瞭であることを示唆します。すなわち、そのPCに沿った変動に負に寄与しています。「x」はPC番号を表します |
seurat_tsne.csv
t-SNEは非線形次元削減法です。2Dまたは3D空間における細胞のクラスターを可視化するために使用されます。ファイルはカンマ区切り形式です。
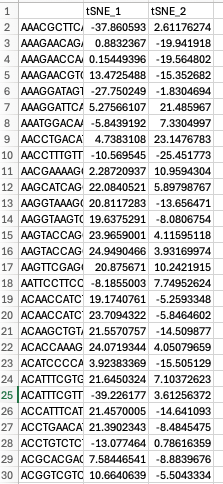
用語
| 用語 | 定義 |
| tSNE_1 | t-SNEプロットの次元1。値はt-SNEプロット上の細胞のx座標を表します。各行は1つの細胞に対応します |
| tSNE_2 | t-SNEプロットの次元2.値はt-SNEプロット上の細胞のy座標を表します。各行は1つの細胞に対応します |
seurat_umap.csv
このファイルには各細胞のUMAP x座標とy座標が含まれています。UMAPは細胞クラスターを可視化するために使用されるもう一つの次元削減技法です。ファイルはカンマ区切り形式です。

用語
| 用語 | 定義 |
| UMAP_1 | UMAPプロットの次元1。値はt-SNEプロット上の細胞のx座標を表します。各行は1つの細胞に対応します |
| UMAP_2 | UMAPプロットの次元2。値はt-SNEプロット上の細胞のy座標を表します。各行は1つの細胞に対応します |
TopListセクションのアウトプットファイル
toplist/toplist/toplist/toplist.zip
このzipフォルダには以下のファイルが含まれています:
| 用語 | 定義 |
| markers_integrated_snn_res.x.json | 0.1-2.0の分解能でサンプル内のクラスターを同定したマーカー遺伝子。「x」はクラスタリングで使用される特定の解像度を表します(json形式) |
| markers_conserved_integrated_snn_res.x.json | 0.1-2.0の分解能で、異なるサンプルグループ間で同定されたクラスターの保存マーカー遺伝子のリスト。「x」はクラスタリングで使用した特定の解像度を表します(json形式) |
| markers_condition_integrated_snn_res.x.json | 0.1~2.0の分解能で、同一クラスター内の異なるサンプルグループ間で発現が異なるマーカー遺伝子のリスト。「x」はクラスタリングで使用した特定の解像度を表します(json形式) |
| heatmap_image_top10_integrated_snn_res.x.png | 0.1-2.0の分解能で同定されたマーカー遺伝子のヒートマップ。「x」はクラスタリングで使用した解像度パラメータを表します(png形式) |
参考文献
1. GitHub. (2024). Single-cell RNA-seq: Marker identification. Github.
関連ブログ
シングルセルRNA-seq解析https://basepairtech.jp/analysis/single-cell-rna-seq/
