CUT&RNA | CUT&TAGの解析ツール
CUT&RUN (Cleavage Under Targets & Release using Nuclease) および CUT&TAG (Cleavage Under Targets & Tagmentation) は、ゲノム全体のタンパク質と DNA の相互作用を高い感度と特異性でマッピングできる次世代シーケンシング (NGS) 技術です。 Basepair は、この種のデータの分析に必要な業界標準ツール (Bowtie2、SEACR、Homer、MACS2 など) へのポイント アンド クリック グラフィカル ユーザー インターフェイスを提供するため、それらを自分で実行する方法を理解するのに時間を費やす必要はありません。
Basepairではじめよう
専用マシンもインストール不要のNGSデータ解析クラウドサービス
BasepairはSaaSです。専用マシンやソフトウェアのインストールは不要です。マシンやPCのスペックを検討したり、たくさんのソフトウェア、プログラムをインストールする際に、バージョンや依存関係の確認なども必要ありません。内部プログラムは実績のあるパブリックなもので構成。論文も多数出版されています。
生データをアップロードしてパイプラインを選ぶだけ
サンプルデータは、FASTQなど生データをアップロードできるほか、Basespaceからの転送、GEO/NCBIからの転送、FTPへの接続にも対応しています。複数のサンプルをアップロードする場合は、APIを使ったコマンドラインでの操作(CLI)にも対応しています。多くのパイプラインはシーケンサーに関係なく解析可能です。
サンプルあたりの従量課金ー初期費用・解析追加料金なし
課金はサンプル数のみに依存する従量制です。アップロードされたサンプルに対する解析は無制限・追加課金ありません。また、初期費用も不要です。
※ 多量のサンプルを取り扱う場合は、年間ライセンスもご用意しています。
業界標準ツールによる透明性と再現性
Basepairは、QC やチャートの読み取りから、すぐに出版できるインタラクティブなプロットに至るまで、再現性と透明性を重視して設計されています。 Basepairで利用できるすべての CUT&RUN ツールと CUT&TAG ツールは、高く引用され、査読されている業界標準のアルゴリズムです。 それらには次のものが含まれます。各種プロットは、PNG、SVGで、各種テーブルはCSVでのダウンロードが可能です。その他の中間ファイルなどもダウンロード可能です。
- Bowtie2
- 高感度と特異性で高品質のリードを参照ゲノムまたはアセンブリに位置合わせする、高速かつ正確なアライメント ツールです。 幅広いリード長を処理できるように設計されており、さまざまな生物や実験条件からの CUT&RUN および CUT&TAG NGS データの分析に適しています。
- SEACR (自己相関領域の信号抽出)
- CUT&RUN および CUT&TAG NGS データ分析用に特別に設計されたツールです。 データからシグナルを抽出し、高い感度と特異性で濃縮された領域を特定します。 SEACR は、そのようなデータに固有の自己相関も補正し、その結果、より正確なピーク コールとダウンストリーム分析の向上が実現します。
- Homer
- ChIP-seq、CUT&RUN、CUT&TAG NGS データを分析するためのツールスイートです。 これには、モチーフの発見、ピークの呼び出し、および差分分析のためのツールが含まれています。 Homer のピーク コーリング アルゴリズムは、動的なバックグラウンド モデルを使用して、ノイズの多いデータであってもピークを正確に識別します。 また、遺伝子オントロジーやパスウェイ分析などの機能アノテーションのためのツールも含まれています。
- MACS2 (Model-based Analysis of ChIP-Seq 2)
- ChIP-seqと =CUT&RUN および CUT&TAG NGS データの両方におけるピーク コール用のもう 1 つの人気のあるツールです。 確率モデルを使用してピークを識別し、ピーク呼び出しとピークフィルタリングのための多数のオプションが含まれています。 MACS2 には、視覚化、注釈、差分分析のためのツールも含まれています。
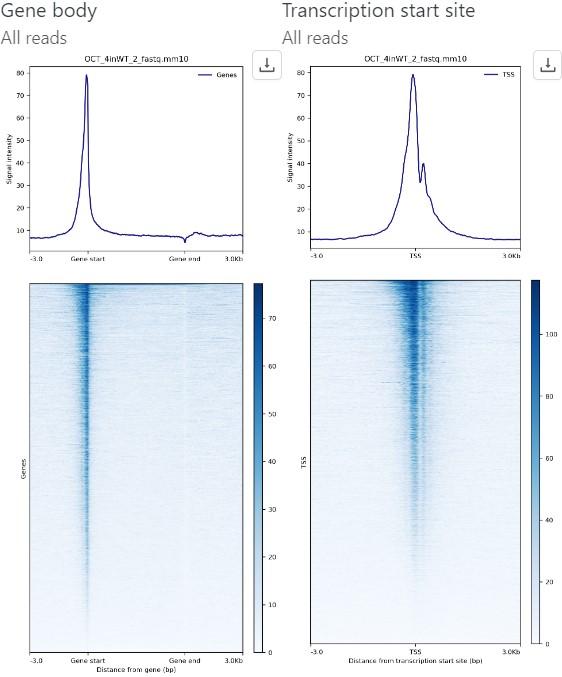
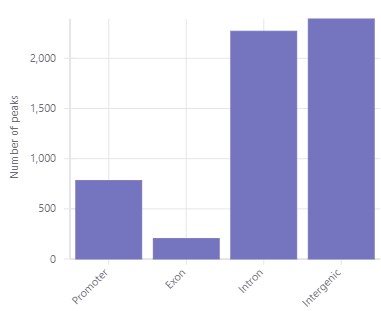
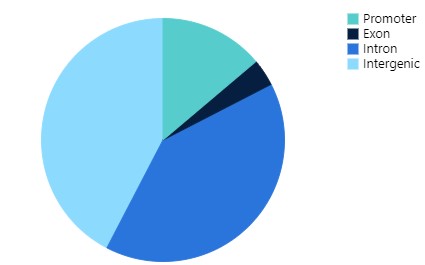
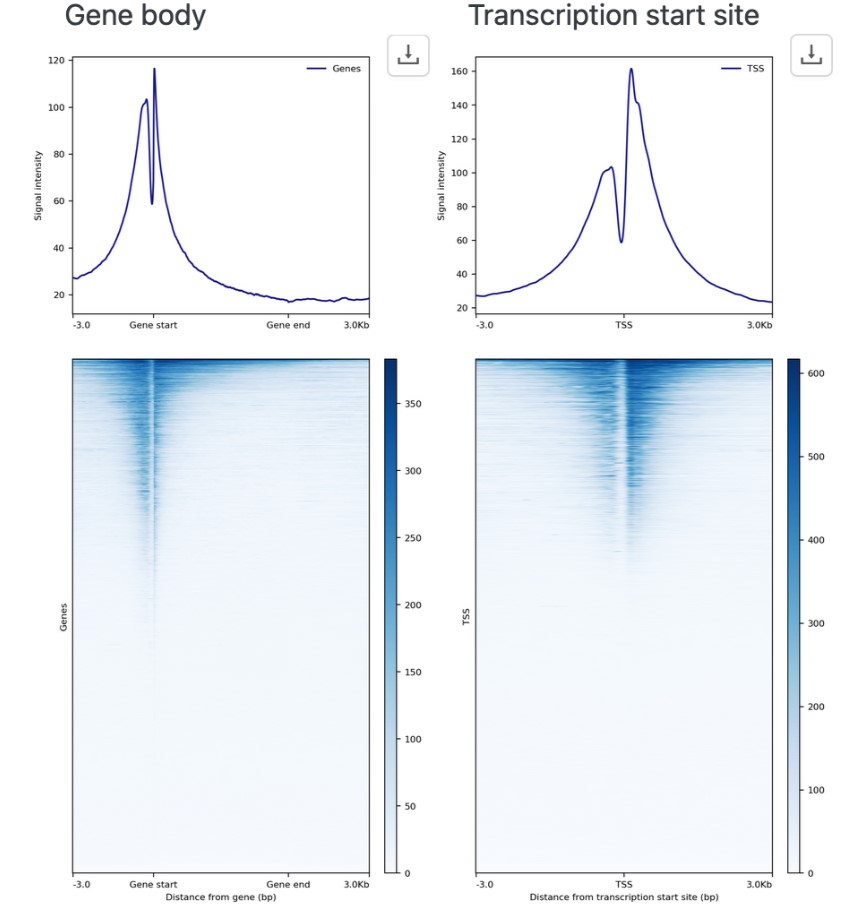
Basepairは、査読ジャーナルにも使われています。
6サンプル フリートライアル 実施中
最大6つのサンプルを無料でアップロードして分析できます。アップロードされたサンプルに対する解析は無制限です。世界トップクラスの機関、研究室、製薬チームがBasepairを使用して、数千ドルを節約している理由をご覧ください。