パイプラインのアウトプットファイルは、「Input/output」タブにあります。
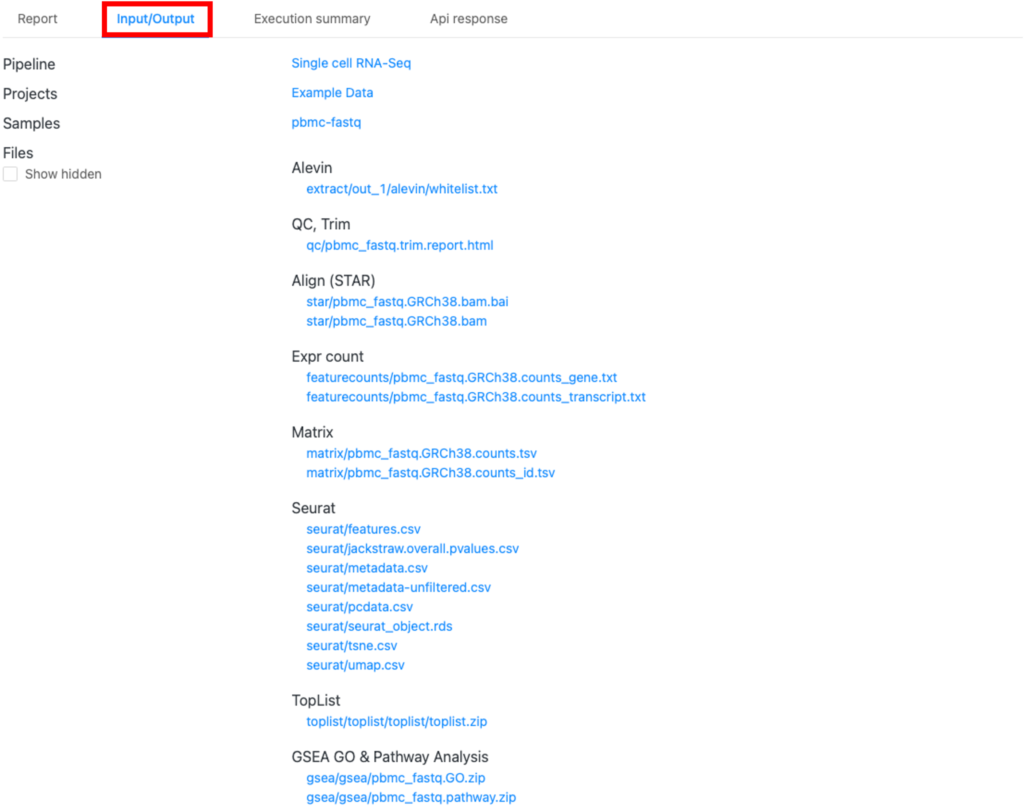
Alevin
| ファイル名 | ファイルの説明 |
| extract/out1/alevin/whitelist.txt | データから特定された有効な細胞のバーコードのリスト。これらのバーコードは、特定の品質閾値超えの個々の細胞に該当します (テキスト形式) |
QC, Trim
| ファイル名 | ファイルの説明 |
| qc/<SAMPLE_NAME>.trim.report.html | 生リードの品質管理およびトリミング後の詳細レポート。リードの品質に関する統計情報が含まれます。例えば、塩基の品質と長さの分布などです (html形式) |
Align (STAR)
| ファイル名 | ファイルの説明 |
| star/<SAMPLE_NAME>.<genome>.bam | STARによって生成された圧縮BAMファイル。リファレンスゲノムにマッピングされたシーケンスデータからのアラインメントリードを含みます |
| star/<SAMPLE_NAME>.<genome>.bam.bai | BAMファイルのインデックスファイル。ゲノムブラウザなどのツールで、BAMファイルへの効率的なランダムアクセスを可能にします |
Expr count
| ファイル名 | ファイルの説明 |
| featurecounts/<SAMPLE_NAME>.<genome>.counts_gene.txt | featureCountsによって生成された遺伝子レベルの発現数。各遺伝子に割り当てられたリードの数を提供します (テキスト形式) |
| featurecounts/<SAMPLE_NAME>.<genome>.counts_transcript.txt | featureCountsによって生成された遺伝子レベルの発現数。遺伝子(テキスト形式)ではなく、個々の転写産物(アイソフォーム)に割り当てられたリードの数を提供します (テキスト形式) |
Matrix
| ファイル名 | ファイルの説明 |
| matrix/<SAMPLE_NAME>.<genome>.counts.tsv | データセット中のセル間で各遺伝子に割り当てられたリードの生カウントを含むマトリックスファイル。行は遺伝子、列は細胞を表します (タブ区切り形式) |
| matrix/<SAMPLE_NAME>.<genome>.counts_id.tsv | counts.tsv ファイルで使用される遺伝子 ID(または転写産物 ID)。遺伝子・転写産物名とカウントデータの対応付けに役立ちます |
Seurat
| ファイル名 | ファイルの説明 |
| seurat/features.csv | Seurat解析で同定された遺伝子のリスト (カンマ区切り形式) |
| seurat/jackstraw.overall.pvalues.csv | SeuratのJackStraw解析によって生成されたp値。JackStrawは、PCAのPrincipal Component(PC)の統計的有意性を決定するために使用されます。どのPCが生物学的に意味あるかを特定します (カンマ区切り形式) |
| seurat/metadata.csv | フィルタリング、正規化、クラスタリング後のSeuratオブジェクトの各細胞のメタデータ。細胞のタイプやクラスタリング結果などの情報が含まれます (カンマ区切り形式) |
| seurat/metadata-unfiltered.csv | データ処理前の各細胞のメタデータ (カンマ区切り形式) |
Seurat
| ファイルの名 | ファイルの説明 |
| seurat/pcdata.csv | SeuratのPCAステップから得られた各細胞のPCスコア (カンマ区切り形式) |
| seurat/seurat_object.rds | RDS(R Data Serialization)ファイル。処理・解析後のSeuratオブジェクト全体を保存しています。生データと正規化されたデータとメタデータが含まれています |
| seurat/tsne.csv | 各細胞のt-SNE (t-distributed Stochastic Neighbor Embedding)座標。t-SNEは非線形次元削減法です。2D・3D空間における細胞のクラスターを可視化するために使用されます (カンマ区切り形式) |
| seurat/umap.csv | 各細胞の UMAP (Uniform Manifold Approximation and Projection)座標。UMAPも細胞のクラスターを可視化する技術です (カンマ区切り形式) |
TopList
| ファイル名 | ファイルの説明 |
| toplist/toplist/toplist/toplist.zip | 同定されたマーカー遺伝子を含むzipファイル。0.1-2.0の解像度のヒートマップも提供します |
GSEA GO & Pathway Analysis
| ファイル名 | ファイルの説明 |
| gsea/gsea/<SAMPLE_NAME>.GO.zip | GOエンリッチメント解析結果を含む圧縮ファイル。各分解能における各クラスターの結果が含まれます |
| gsea/gsea/<SAMPLE_NAME>.pathway.zip | Reactomeのエンリッチメント解析結果を含む圧縮ファイル。各分解能における各クラスターの結果が含まれます |
参考文献
1. GitHub. (2024). scRNA-seq data analysis tools. Github. https://github.com/mdozmorov/scRNA-seq_notes
関連ブログ
シングルRNA-seq解析https://basepairtech.jp/analysis/single-cell-rna-seq/
エンリッチメント解析https://basepairtech.jp/blog/1973/
