はじめに
多くの研究者がGEO(Gene Expression Omnibus)からインポートされたデータを用いて、様々な解析や検討を行っています。しかしながら、関連するメタデータなどを抽出し処理することは、手間のかかる作業です。そこで、BasepairではImport data from GEO というパイプラインを用意しています。
GEOの概要
GEOは、NCBIが支援するインターネット一般公開データベースです。マイクロアレイ、NGSなどの機能ゲノミクスデータがアーカイブされています。現在、世界72カ国以上の研究者から提出されており(1)、6,000以上の生物に700万以上の生物サンプルのデータがアーカイブされています(5)。また、多くのジャーナルや資金提供機関は、GEO上での共有することを研究者に求めています(6)。これは、研究結果の検証を可能にし、データ資産へのアクセスを保証するためです。
データセットは、二次研究や、開発・評価手法に利用することができます(2、3、7)。
たとえば、
- 現在利用可能な解析プラットフォームの互換性を最大化
- 解析アルゴリズムの有効性を比較
- 発現量の異なる遺伝子の同定や疾患発症に影響する経路の同定
- 複雑な生物学的プロセスや疾患に対する洞察
解析のためのGEOデータインポートの課題
GEOのデータセットの複雑さは、以下の複雑さに起因すると考えられています。このため、データアクセスや検索、統合、解析における課題となる可能性があります(4)。
- 多様なデータタイプ
- 実験のバリエーション
- メタデータの複雑さ
データアクセスと取得
GEOには、膨大なデータセットがあり、インターフェースが複雑です。GEOでデータを効率的に探すには、フィルタリングを慎重に検討する必要があります。うまくフィルタリングできた場合でも、アノテーションなどが欠落している場合があります。この場合、GEOのデータのインポートが困難になることがあります。また、ダウンロードができたとしても、解析が困難になる場合があります。そのため、関連するすべてのデータセットをダウンロードし、データセットを相互検証する必要があるかもしれません。これには時間がかかり、NGSデータ解析の経験が浅い研究者には困難が伴います。
データの統合と解析
GEOに含まれる実験プラットフォーム、プロトコール、および生物学的システムの多様性を考慮すると、そのデータセットはしばしば、データの質や前処理方法において大きなばらつきを示します。このばらつきは、サンプル調製、ハイブリダイゼーション技術、バッチ効果、実験条件などの違いから生じます。さらに、一貫性のないメタデータ、不十分なメタデータの提出、GEOデータセット上の注釈の欠落などに起因するメタデータの複雑さも起こりうります。異種のGEOデータセットとメタデータの複雑さは、結果的に解析ツールでのデータ統合を妨げ、特にバイオインフォマティクススキルのない研究者にとっては下流の解析を複雑にします。
解析に先立ち、ユーザーはGEOデータセットのメタデータと注釈フィールドを抽出、解釈、処理する必要があります。場合によっては、データ解析の前に、これらのフィールドを整理したり再構築したりして、データを解析ツールに適した使用可能な形式にする必要があり、そのためにはコーディングスクリプトを作成する必要があります。これらは、特に実験用語に不慣れであったり、コーディングスキルが不足していたりする研究者にとって、困難な場合があります4。
BasepairへのGEOデータのインポート
BasepairのImport data from GEOは、二次分析に必要なデータセットを、簡単に直接GEOからBasepairにインポートすることができます。必要なインプットは、解析したいデータセットのアクセッション番号のみです。また、複数のデータセットを同時にインポートすることもできます。
Basepairにデータがインポートされると、プロジェクト内に保存されます。その後、インポートされたサンプルの解析が実行されると、解析パイプラインが実行に必要なメタデータやアノテーションを抽出、統合します。
簡単なワークフロー
- Basepairのアカウントにログインしてください。
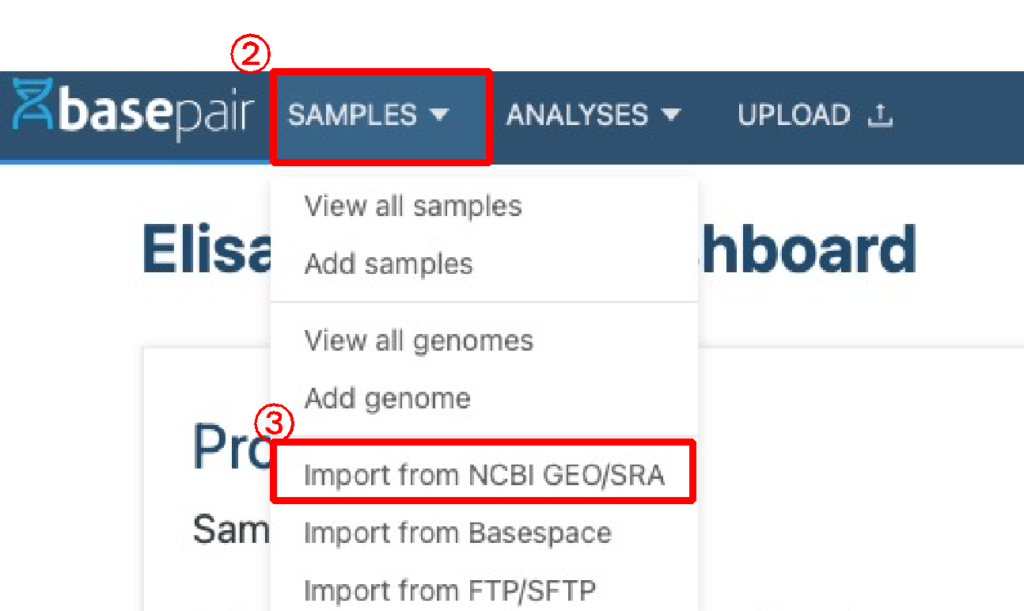
- 「SAMPLES 」をクリックしてください。
- 「Import from NCBI GEO/SRA」をクリックしてください。
- 「Import data from GEO」パイプラインを選択してください。
- データのGEOまたはSRAのアクセッションID を入力してください。複数のIDを入力することもできます。
- GEOアクセッション ID例:GSM1382485
- SRAアクセッション ID例:SRX513011
- 必要に応じて「Analysis name」は変更できます。
- 「Run analysis」をクリックしてください。
- インポートが完了すると、サンプルはBasepair上のプロジェクトに保存されます。

留意点
GEOからのデータのインポートは、多くの場合スムーズに行われます。しかしながら、以下のようなケースが時々起こりますので、ご注意ください。
- そもそもRaw Dataが公開されていない
- Data Typeが間違っている
- 例)シングルセルなのに、RNA-Seqとなっている
- Basepair上で、データタイプを変更することで解決します
- メタデータが間違っている
- 例)シングルセルのUMIがついていない
- データがdbGaPにある
- Basepairからはアクセスできません
GEOへのデータのコピー
Basepairには、GEOへのデータコピーのパイプラインも用意しています。併せてご覧ください。
参考文献
- Clough, E., Barrett, T. (2016). Methods Mol Biol., 1418: 93-110.
- Eren, K., Deveci, M., Küçüktunç, O., et al. (2013). Briefings Bioinf, 14:279–92.
- Golightly, N.P., Bell, A., Bischoff, A.I., et al. (2018). Sci Data, 5:180066.
- Mecham, A., Stephenson, A., Quinteros, B.I., et al. (2023). J Integr Bioinform, 20230021.
- NCBI (2024). NCBI.
- Wilkinson, M. D., Dumontier, M., Aalbersberg, I.J., et al. (2016). Sci Data., 3:160018.
- Zhou, W., Han, L., Altman, R.B. (2016). Bioinformatics, 33:522–8.
