シングルセルシーケンスの概要
シングルセルシークエンシングは、単一細胞レベルのオミクス分野における新時代の到来を告げています[1]。 すべての細胞は機能において独自であり、システムの生物学を定義する明確な系統を持っています。 シングルセル解析は、細胞間のゲノムおよびトランスクリプトームレベルの違いを解読し、生体サンプルの不均一性を明らかにするプロセスです。 多細胞生物から単離された個々の細胞のユニークな特性を明らかにすることで、医学、発生生物学、システム生物学の分野に変革をもたらしてきました [2、3] 。 シングルセル解析では、各細胞のプロファイルを生成して、部分集団内および部分集団間の差異を特定します。 これにより、同じ刺激に対する個々の細胞の反応の特徴付けが可能になり、個々の細胞の表現型とそれらが生物の全体的な機能にどのような影響を与えるかについての知識が高まります。
生体サンプルは本質的に不均一です。 たとえば、腫瘍生検のために細胞を収集する場合、健康な細胞と癌性の細胞の両方が存在することがよくあります。 NGS バルクシーケンスの従来の方法では、サンプル内のすべての細胞タイプのデータが混合されます。 したがって、バルク細胞 NGS 分析では、正常細胞と腫瘍細胞から発せられるシグナルを常に容易に識別できるわけではなく、異なる腫瘍細胞部分集団を区別することもできません。 シングルセルでは、各細胞の DNA/RNA に固有の DNA バーコードを組み込むことでこの問題を解決します。 細胞が追加されると、細胞はプールされ、一緒に配列決定されます。 シーケンスが正常に完了すると、バーコードの存在に基づいて各セルの読み取りを簡単に分離できます。
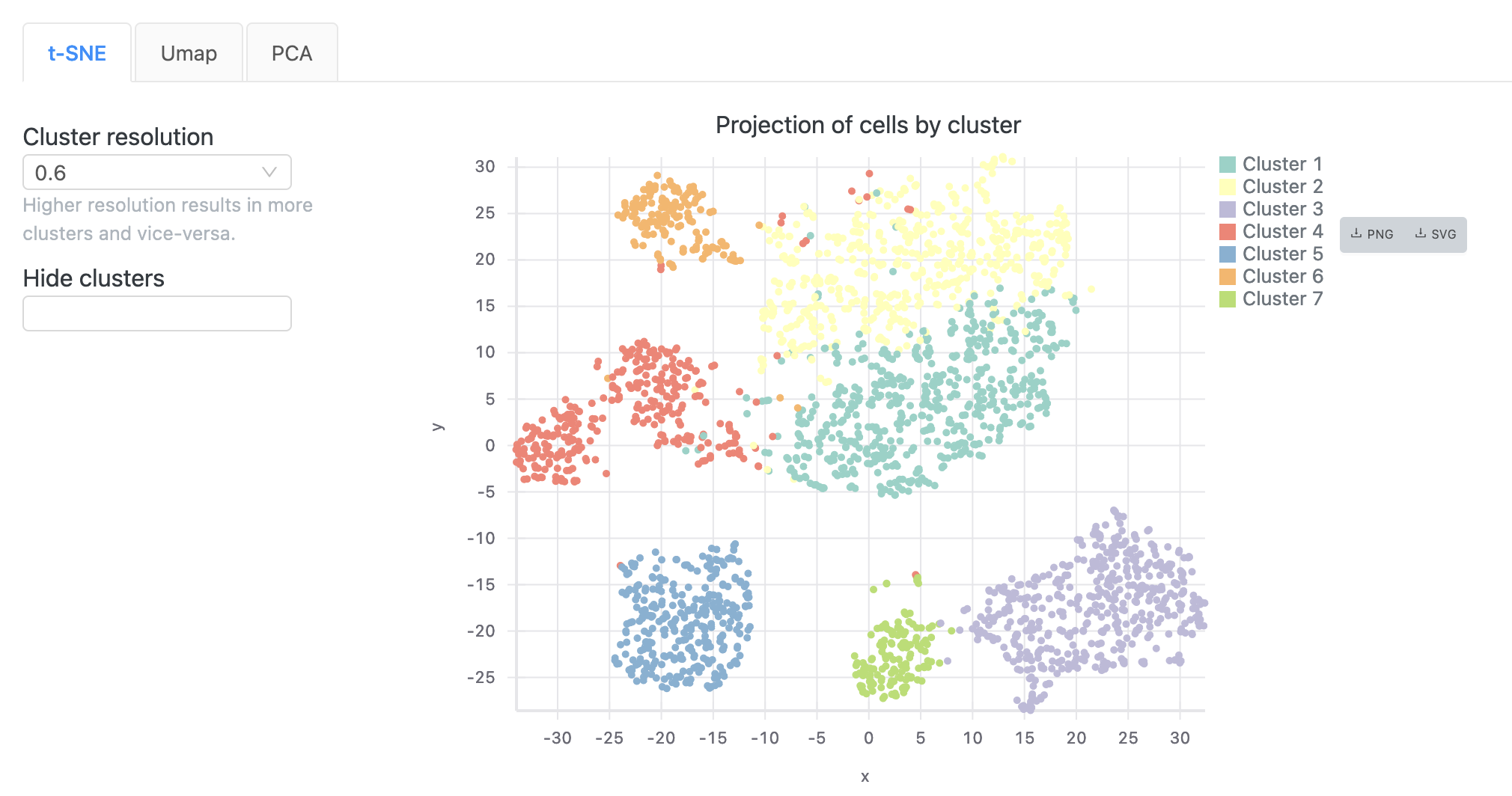
これまでに、シングルセル解析、クラスタリング、系統解析などの多くの手法が開発されてきました。 たとえば、Basepair のシングルセルRNA-seq(scRNA-Seq) パイプラインは、シングルセルRNA-seq データを分析するために設計された一般的なツールである Seurat を使用します。 これは、研究者が細胞部分集団を特定して視覚化し、各部分集団に固有の遺伝子マーカーを特定し、複数の scRNA-seq データセットをマージするのに役立ちます。 クラスタリング法は、細胞とその遺伝子発現プロファイルの間の関係を捉える細胞間類似性測定を通じて細胞集団を特定するのに役立ちます。
現在の課題と将来の展望
シングルセル解析は急速に出現した技術ですが、大幅な進歩にもかかわらず、多くの重大な課題が依然として残っています。 シングルセルシークエンシングの主な課題の 1 つは、微量の DNA または RNA から多様なゲノム、トランスクリプトーム、およびエピジェネティックなイベントを確実に検出することです。 研究者が直面する一般的な問題は、生存細胞と死滅または瀕死の細胞の違いを見分けること、または細胞ではなく周囲/汚染された DNA または RNA の配列を決定することです。
その他の課題としては、配列決定のコストと、膨大なデータセットを扱うためのバイオインフォマティクス ソフトウェアの開発が挙げられます。 通常、シングルセルシーケンスは、従来のバルク NGS シーケンスと比較して時間がかかり、コストも高くなります。 シングルセルデータセットから有意義な結果を抽出するための自動化とバイオインフォマティクス技術の開発も緊急に必要とされています。 シングルセル解析に利用可能なアルゴリズムとソフトウェア パッケージは、標準的なバルク NGS シーケンスに比べて成熟度が低いです。 単一シーケンス、特にシングルセルRNA-seq によって生成されるデータは、バルク RNA-seq データよりもノイズが多く、大規模な計算インフラストラクチャを必要とするため、データ分析は非常に要求が厳しくなります。 これらの課題を克服し、包括的で自動化されたシングルセル解析ツールに対する研究者のニーズを満たすために、Basepair は シングルセル RNA-seq 解析 パイプラインを作成しました。
シングルセル解析の応用
シングルセルは、がん研究、幹細胞生物学、発生生物学、その他の分野において非常に重要です [4、5] 。 単一細胞の配列決定と解析は、根底にある生物学的プロセスを前例のない解像度で明らかにすることで、細胞生物学の分野に革命をもたらしました [6] 。 癌性幹細胞の分子プロファイリングは、転移の根本的な原因や多くの治療反応を発見するのに役立ちました[7]。 シングルセルは、ゲノムの配列を決定することによる日常生活における細菌種の重要性のさらなる解明から、神経細胞の分析による神経発達と中枢神経系の動態の解明に至るまで、多くの重要な疑問を解決する可能性を秘めています[8]。 さらに、疾患のプロセスと関連する治療介入を理解するために不可欠な新規薬剤標的の同定を可能にすることで、医学分野における最先端の応用を提供します。
BasepairのシングルセルRNA-Seq
BasepairのシングルセルRNA-Seqは、fastqをアップロードするだけで、QC、アライメントから視覚化まで一気に完了します。
References
- Wang, D., & Bodovitz, S. (2010). Single cell analysis: the new frontier in ‘omics’. Trends in biotechnology, 28(6), 281-290.
- Chi, K. R. (2013). Singled out for sequencing: Nature Publishing Group.
- Eberwine, J., Sul, J.-Y., Bartfai, T., & Kim, J. (2014). The promise of single-cell sequencing. Nature methods, 11(1), 25.
- Kato, T. (1980). Biochemical characteristics of individual neurons Advances in cellular neurobiology (Vol. 1, pp. 119-176): Elsevier.
- Li, X., & Lee, A. P. (2018). High-throughput microfluidic single-cell trapping arrays for biomolecular and imaging analysis. Methods in cell biology, 148, 35-50.
- Benitez, J. A., Cheng, S., & Deng, Q. (2017). Revealing allele-specific gene expression by single-cell transcriptomics. The international journal of biochemistry & cell biology, 90, 155-160.
- Baslan, T., & Hicks, J. (2017). Unravelling biology and shifting paradigms in cancer with single-cell sequencing. Nature Reviews Cancer, 17(9), 557.
- Ofengeim, D., Giagtzoglou, N., Huh, D., Zou, C., & Yuan, J. (2017). Single-cell RNA sequencing: unraveling the brain one cell at a time. Trends in molecular medicine, 23(6), 563-576.