パスウェイ解析とは
Basepairではパスウェイ解析も行うことができます。あらかじめ定義された遺伝子や遺伝子産物の位置、遺伝子同士の相互作用などの情報を含んだモデルをもとに行うため、より生物学的な議論を行うために重要な情報を提供してくれます。
パスウェイ解析は次のような場合に役立ちます
- 測定された遺伝子発現変化の大きさと方向を理解したい。
- 全遺伝子の様々な位置と役割、およびパスウェイ上の遺伝子間の相互作用を理解したい。
- 下流またはパスウェイレベルの効果を理解したい。
- 例えば、遺伝子産物または単一の受容体がどのようにパスウェイをトリガーするか。
パスウェイと遺伝子セットの違い
遺伝子発現解析の際、パスウェイ解析を行うことでサンプルの背景にある生物学的プロセスやメカニズムをより深く理解するために行います。GSEA(エンリッチメント解析)には遺伝子セット解析とパスウェイ解析があります。
- 遺伝子セット:遺伝子の秩序化されていないコレクション
- パスウェイ:与えられた生物学的プロセス、メカニズム、現象を記述する複雑なモデル
MAPKの例
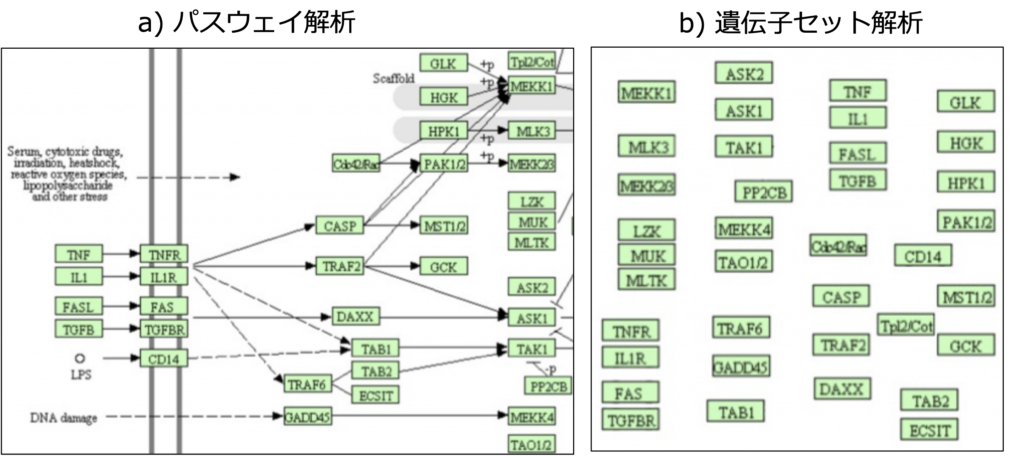
例として、図1ではKEGG MAPKパスウェイ(図1a)とKEGG MAPKパスウェイに対応するMSigDB遺伝子セット(図1b)を比較したものです。パスウェイでは、様々な遺伝子や遺伝子産物の位置(細胞内、細胞外、膜内など)、遺伝子同士の相互作用、それぞれの相互作用の種類(活性化、抑制など)、シグナルの伝播方向などが示されています。一方、遺伝子セットは、元のパスウェイが捉えていた構造や重要な情報をすべて失っています。
つまり、パスウェイ解析は、遺伝子セット解析と比較して、パスウェイによって描写される生物学的プロセスに関するより多くの情報を得ることができます。
BasepairのDESeq2パイプラインに実装されています。これらは互いに並行して実行されます。
Basepairでのパスウェイ解析
BasepairのDESeq2パイプラインでは、GSEAまで完了します(図2)。
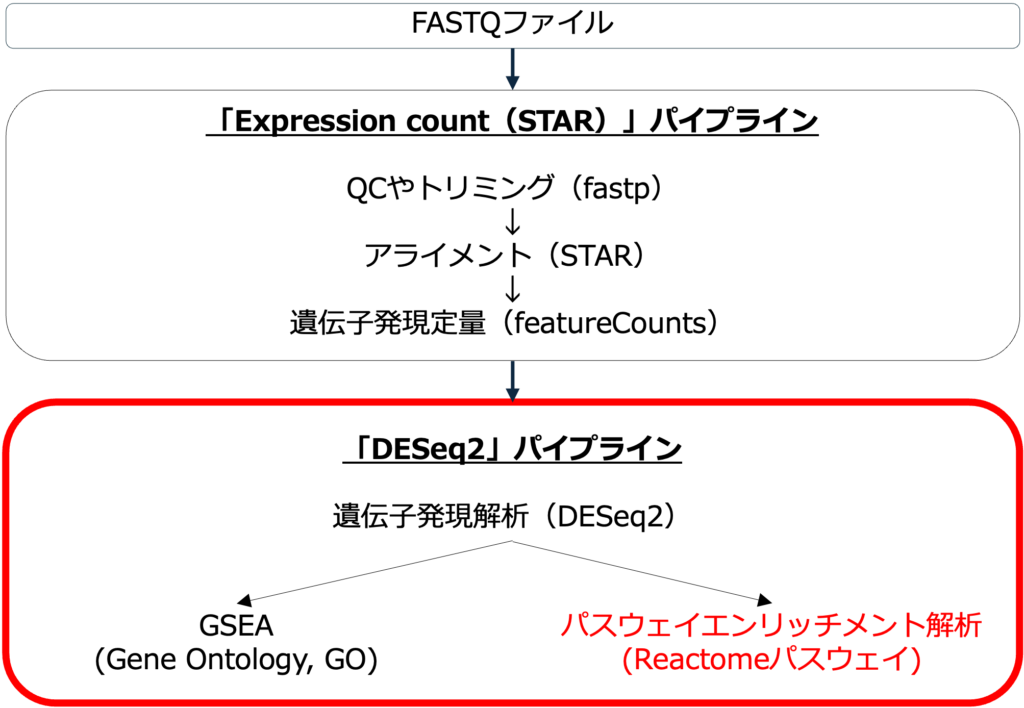
インプット
FASTQファイル
ステップ
- 「DESeq2」パイプラインを選択します。
- 「Expression count (STAR)」パイプラインが「DESeq2」パイプラインの前に自動的に実行されます。ここでは、品質管理(QC)、データトリミング、アライメントが行われます。
- DESeq2パイプラインが開始され、発現変動解析が実行されます。ここでは、後段の解析のために以下の3つのファイルが生成されます:
- 正規化された遺伝子発現レベルを含むタブ区切りファイル (norm.gct)
- norm.gctファイル中のサンプルのクラスを含むクラスファイル(cls)
- norm.gctファイル中の識別子を遺伝子セットファイル(gmt)中の識別子にマッピングしたChipファイル(chip)
- Reactomeパスウェイデータベースから必要なパスウェイ遺伝子セットファイル(gmt)をMolecular Signatures Database (MSigDB)から自動的に抽出します。
- 発現変動遺伝子解析が完了すると、GOエンリッチメント解析とパスウェイエンリッチメント解析が自動的に並行して実行されます。
DESeq2パイプラインを実行すると、Expression count (STAR)が自動的に実行されます。Basepairが自動で行うので、ファイルを用意する必要はありません。
レポート
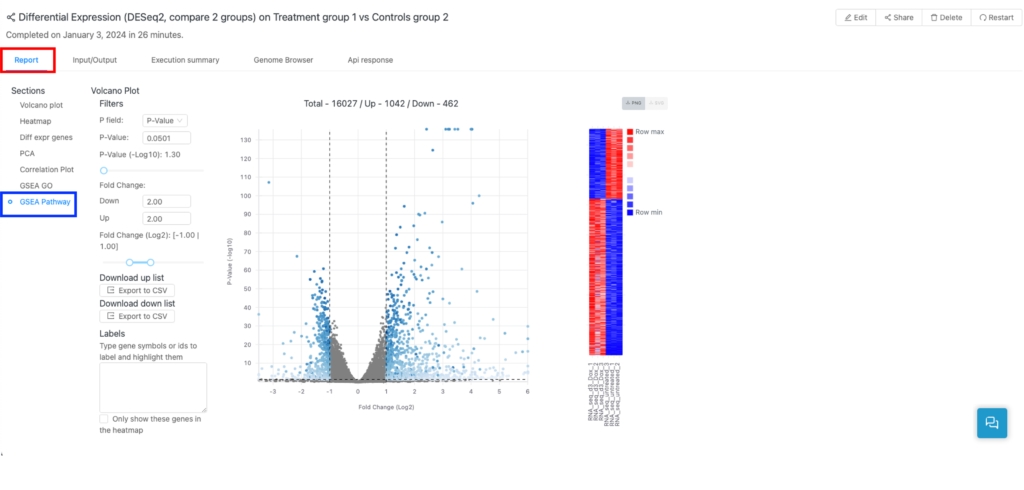
発現変動遺伝子(DEGs)の有意にエンリッチメントされたパスウェイの結果は、「Report」タブの下にある「GSEA Pathway」セクションのインタラクティブな表で示されます(図3)。
有意にエンリッチメントされた DEG のパスウェイとその統計量を図 4 に示します。この例では、「Treatment_group_1」タブでエンリッチメントされたパスウェイは、発現が増加したDEGのパスウェイを表し、「Control_group_2」タブでエンリッチメントされたパスウェイは、発現が減少したDEGのパスウェイを表します。
有意にエンリッチメントされた DEG のパスウェイとその統計量を図 4 に示します。この例では、「Treatment_group_1」タブでエンリッチメントされたパスウェイは、発現が増加したDEGのパスウェイを表し、「Control_group_2」タブでエンリッチメントされたパスウェイは、発現が減少したDEGのパスウェイを表します。
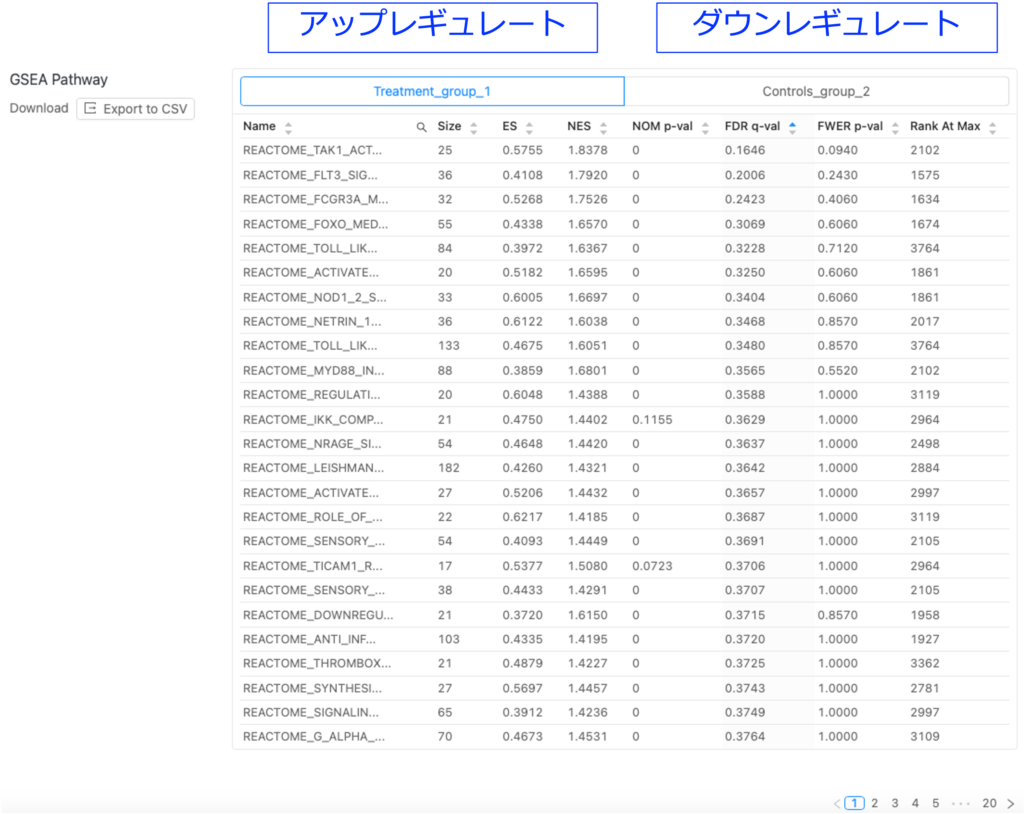
エンリッチされたパスウェイの表(図4)で使われている用語
| Name | 事前に定義されたパスウェイ遺伝子セットの名前。 |
| Size | 定義された遺伝子セットの遺伝子数。 |
| ES | Enrichment score。ランク付けされたパスウェイ遺伝子リストの上位または下位で、定義された遺伝子セットがどのエンリッチしているかを反映するために使用されるスコア。 |
| NES | Normalized enrichment score。ESをパスウェイ遺伝子セットのサイズのばらつきで正規化したもの。 |
| NOM p-value | Nominal p-value。あらかじめ定義された1つの遺伝子セットのESの統計的有意性。 |
| FDR q-value | False discovery rate q-value。所定のNESを持つ遺伝子セットが偽陽性である確率の推定値。 |
| FWER q-value | Family-wise error rate。結果のリストに1つも偽陽性パスウェイ遺伝子セットが含まれないことを保証するための統計的有意性のより保守的な推定値。 |
| Rank at max | ランク付けされたリストの中で、ESが最大となった位置。 |
まとめ
パスウェイエン解析は、遺伝子セット解析と比較して、より多くの情報を提供します。Basepairでは、パスウェイエンリッチメント解析をGSEAを並行して実行します。
参考文献
最大6サンプル フリートライアル 実施中
最大6つのサンプルを無料でアップロードして分析できます。アップロードされたサンプルに対する解析は無制限です。世界トップクラスの機関、研究室、製薬チームがBasepairを使用して、数千ドルを節約している理由をご覧ください。
