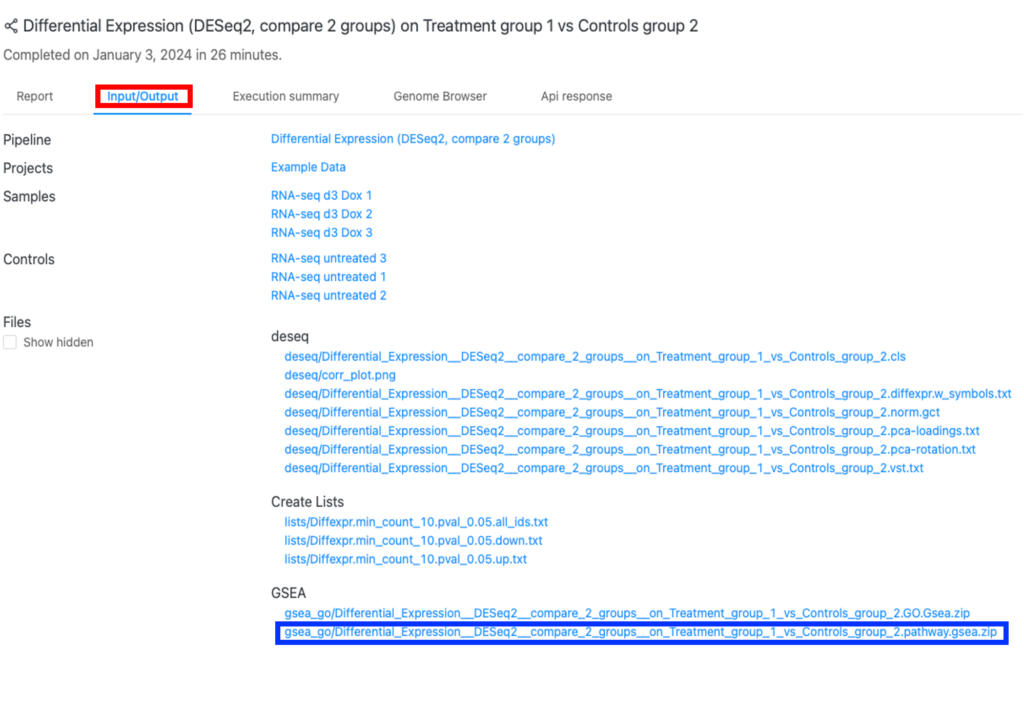
DESeq2パイプラインでパスウェイエンリッチメント解析を実行するとGSEAレポートが作成されます。このレポートはzipフォルダ(図1の青枠)にあります。このフォルダは「Input/output」タブ(図1の赤枠)からアクセスできます。このレポートには、エンリッチされたパスウェイに関する情報が含まれています。
GSEAレポート(index.html)
GSEAのレポートはhtml形式です(図2)。レポートには6つのセクションがあります:
- Enrichment in phenotype 1
- Enrichment in phenotype 2
- Dataset details
- Gene set details
- Gene markers
- Global statistics and plots

「Enrichment in Phenotype」セクション
「Enrichment in Phenotype 」には、2つのセクションがあります。最初のセクションでは、ポジティブのES(Enrichment Score、エンリッチメントスコア)のパスウェイの結果を示します。すなわち、ランク付けされたリストの上位でエンリッチメントを示すパスウェイです。2つ目のセクションでは、ネガティブのESを持つパスウェイの結果を示します。つまり、ランク付けされたリストの下位でエンリッチメントを示すパスウェイです。図2では、表現型1がトリートメントグループ、表現型2がコントロールグループです。

各表現型の情報
| 情報 | 図中の例 |
| 表現型にエンリッチされたパスウェイの数と解析された遺伝子セットの総数 | 485 / 884 gene sets are upregulated in phenotype 1 |
| 有意にエンリッチされたパスウェイの数。これは25%未満のFDR(False discovery rate、偽発見率)によって示されます。FDRは有意な結果の集合の中の偽陽性の割合を推定します | 3 gene sets are significant at FDR < 25% |
| 公称p値が1%未満および5%未満のエンリッチされた遺伝子セットの数 | 140 gene sets are significantly enriched at nominal pvalue < 1% |
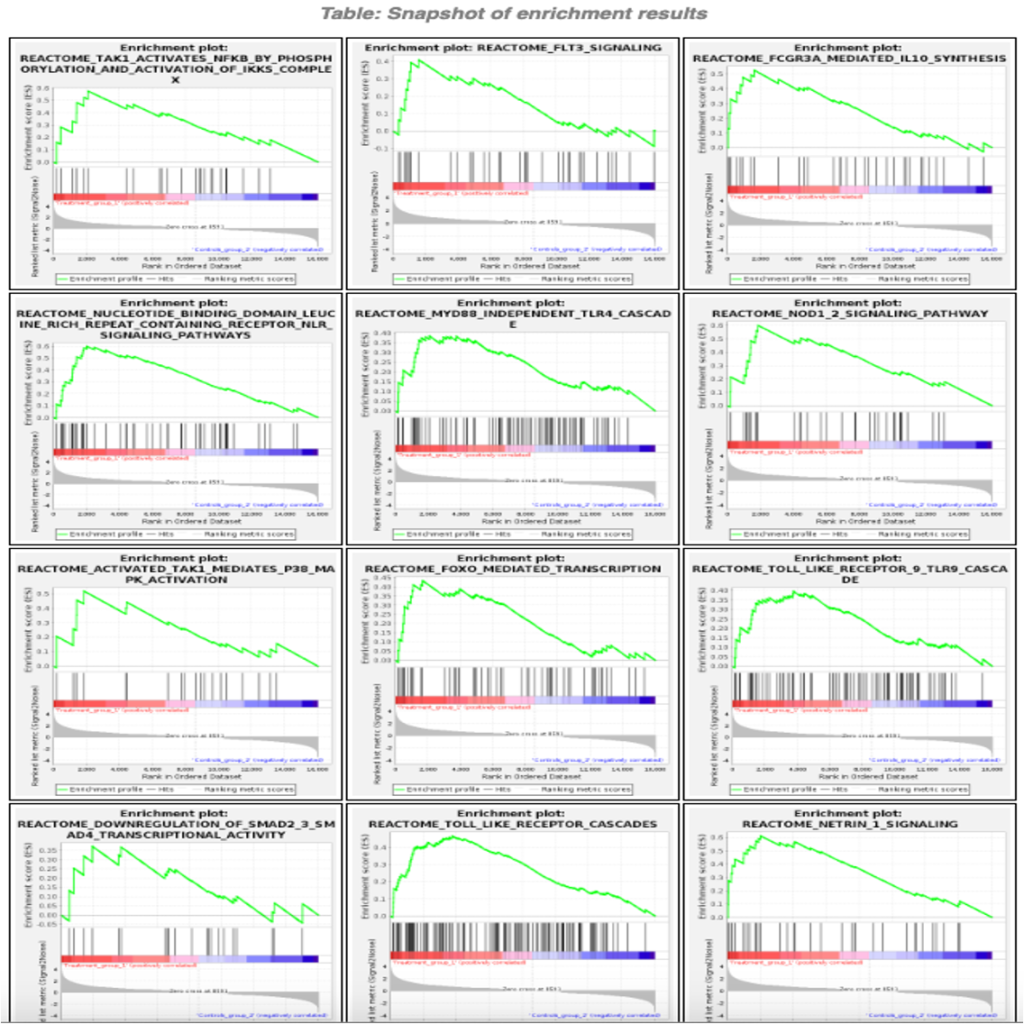
| トップ結果のスナップショット。正規化ESの絶対値が最も高かった遺伝子セットの濃縮プロットが表示されている。 | Snapshot of enrichment results (Figure 3) |
| 表現型にエンリッチされた遺伝子セットの要約レポートを含む詳細なエンリッチメント結果。これらの結果はhtmlとexcel形式です | Detailed enrichment results in html format (Figure 4) |
| このセクションで表示される結果の解釈方法を説明するガイド。MSigDB(1)が提供しています | Guide to interpret results |
Snapshot of enrichment results
「Snapshot」(赤枠)から、結果のスナップショットが確認できます(図3)。

Detailed enrichment results
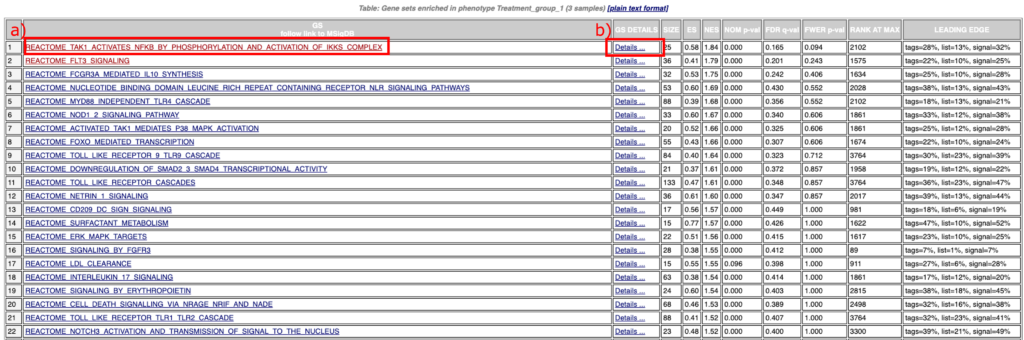
「enrichment results」(赤枠)からリストを確認できます(図4)。ここでは、htmlかtsv形式が選択可能です(赤枠)。

用語
| 用語 | 定義 |
| GS | パスウェイ 遺伝子セット名 |
| GS details | パスウェイ遺伝子セット詳細レポート表示へのリンク |
| Size | 発現データセットに含まれない遺伝子をフィルタリングした後の遺伝子セット内の遺伝子数 |
| ES | Enrichment Score (ES)。遺伝子セットがランク付けされた遺伝子リストの上位または下位にどれだけ強くエンリッチされているかの尺度 |
| NES | Normalized Enrichment Score (NES)。パスウェイ遺伝子セットのサイズに対して生のESを調整します。これは異なるサイズの遺伝子セット間の比較を可能にします |
| NOM p-val | 名目上のp値。観測されたESの統計的有意性を推定します |
| FDR q-val | False Discovery Rate q値。結果のリストに1つの偽陽性遺伝子セットが含まれないように名目上のp値を調整します |
用語
| 用語 | 定義 |
| FWER p-val | Family-Wise Error Rate p値。FDR q-valueよりも統計的有意性を保守的に推定します。偽陽性の確率をコントロールすることで名目上のp値を調整します |
| Rank at max | ランク付けされた遺伝子リストの中で、ESが最大となった位置 |
| Leading edge | リーディングエッジサブセットを定義するために使用された3つのメトリクスを表示します。遺伝子発現の解析に使用された3つの指標を表示します – Tags:ランク付けされた遺伝子リスト全体からの遺伝子の割合 – List:ランク付けされた遺伝子リストのうち、リーディングエッジサブセットに含まれる遺伝子の割合 – Signal:エンリッチメントシグナルに起因するESの割合 |
図4の「Gene Set(GS)」列
GS列(図4a)で詳細なReactomeパスウェイを確認する手順は以下です:
- GS列(図4a)からエンリッチされたパスウェイをクリックします。
- ブラウザが MSigDB 上の実際のパスウェイを表示します(図 5)。
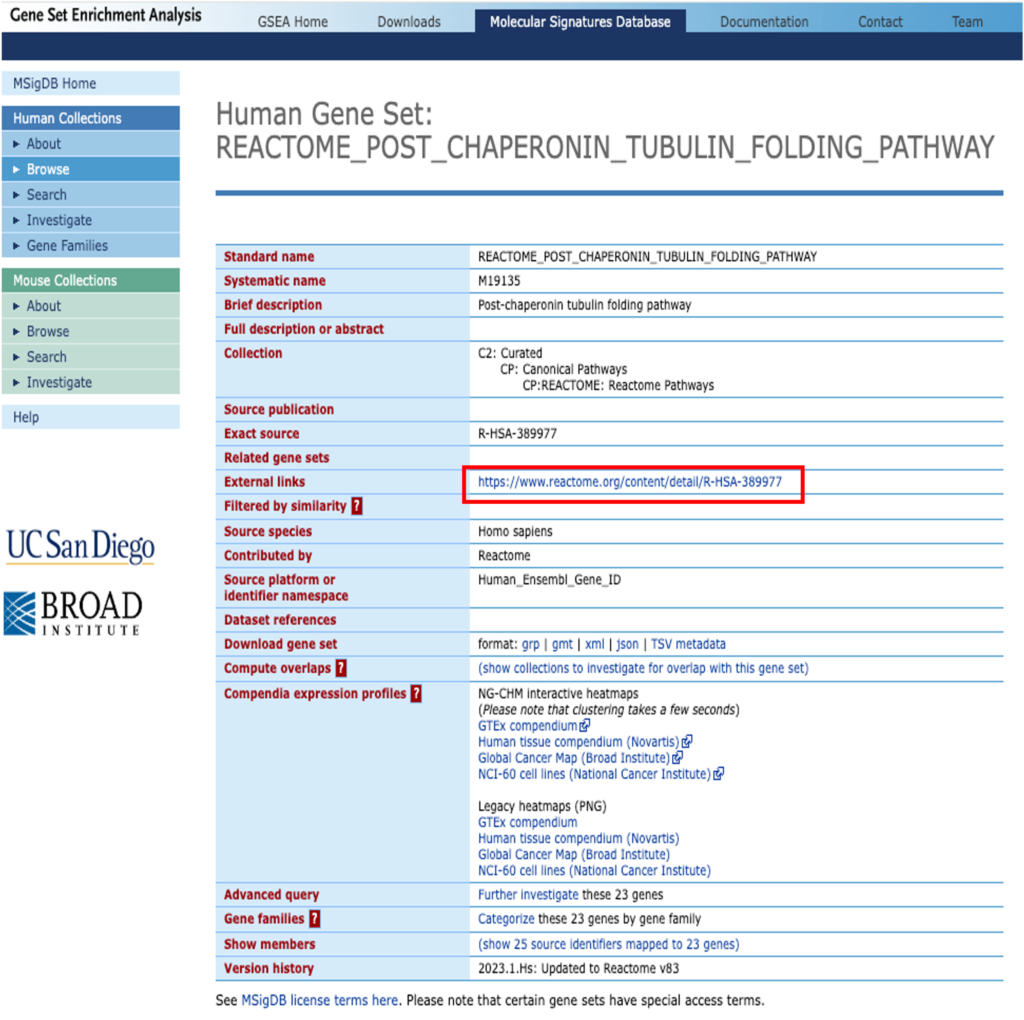
3. 外部リンク(図 5 の赤枠)をクリックすると、Reactome ウェブサイトでパスウェイ全体を詳細に見ることができます(図 6)。
図 4 の「Gene Set(GS)Details」列
パスウェイの詳細を見たい場合は、「Details…」をクリックします(図 4b)。このレポート(図 7)には以下が含まれます:
- エンリッチメント統計量を含むパスウェイのサマリー
- パスウェイのエンリッチメントプロット
- パスウェイに関連する発現変動遺伝子のリスト
- パスウェイの遺伝子の発現レベルを示すヒートマップ
- ランダムES分布プロット
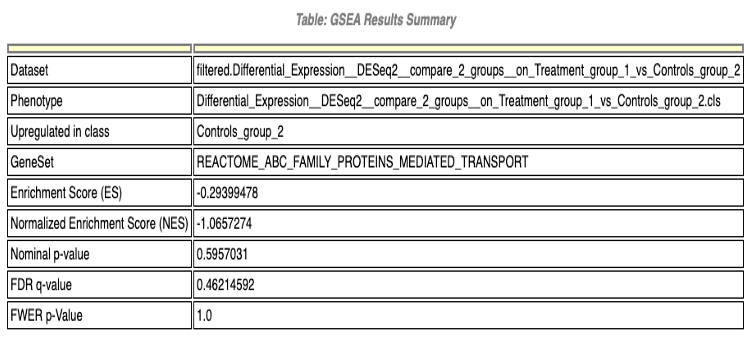
エンリッチメント統計量を含むパスウェイのサマリー
用語
| 用語 | 定義 |
| Dataset | 解析対象データセット |
| Phenotype | パスウェイエンリッチメント解析で比較される生物学的条件 |
| Upregulated in class | パスウェイがエンリッチされる表現型 |
| GeneSet | Reactomeデータベースで生物学的機能に基づいてグループ化された遺伝子のコレクション |
| Enrichment score (ES) | データセット中の遺伝子セットがどの程度エンリッチされているかを反映するスコア |
| Normalized Enrichment Score (NES) | 遺伝子セットのサイズの違いを考慮して正規化されたES |
| Nominal p-value | 名目上のp値。遺伝子セットのESの統計的有意性を推定する値 |
| FDR q-value | False Discovery Rate (FDR) q値は有意な遺伝子セットの中で偽陽性を考慮してp値を調整します |
| FWER p-value | Family-Wise Error Rate (FWER) p値は、FDR q値よりも保守的な多重検定の補正値で、エンリッチされた遺伝子セット間の偽陽性を考慮します |
パスウェイのエンリッチメントプロット
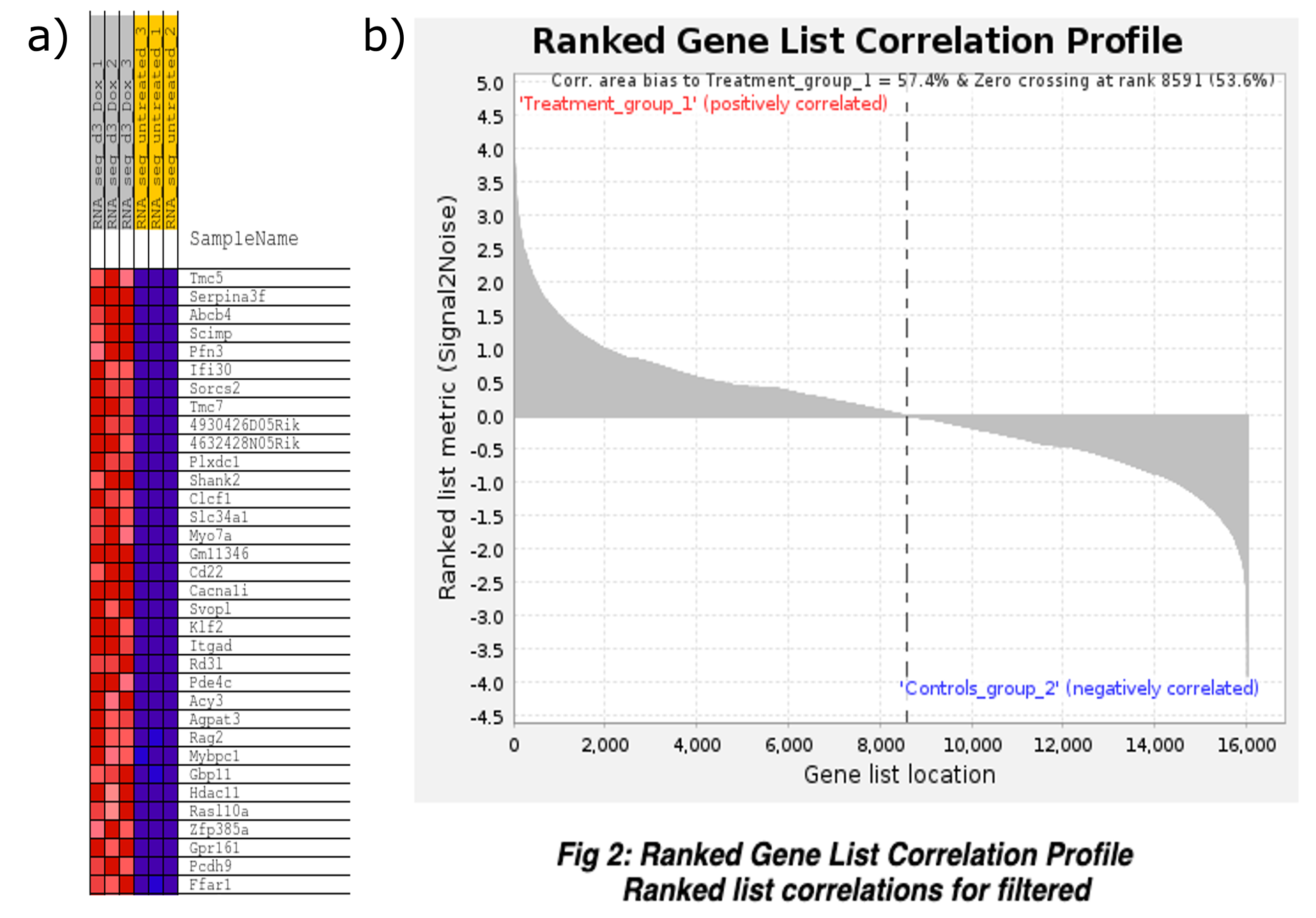
図8はパスウェイのエンリッチメントプロットです。このプロットでは、あるパスウェイからの遺伝子が、ランク付けされた遺伝子リストにどのように分布しているかを示しています。さらに、パスウェイからの遺伝子がどの程度エンリッチされているかを示します。図8には、a) ESプロファイル、b) 遺伝子セットメンバーの位置、c) ランク付けされたリストの相関プロファイルの3つのセクションがあります。
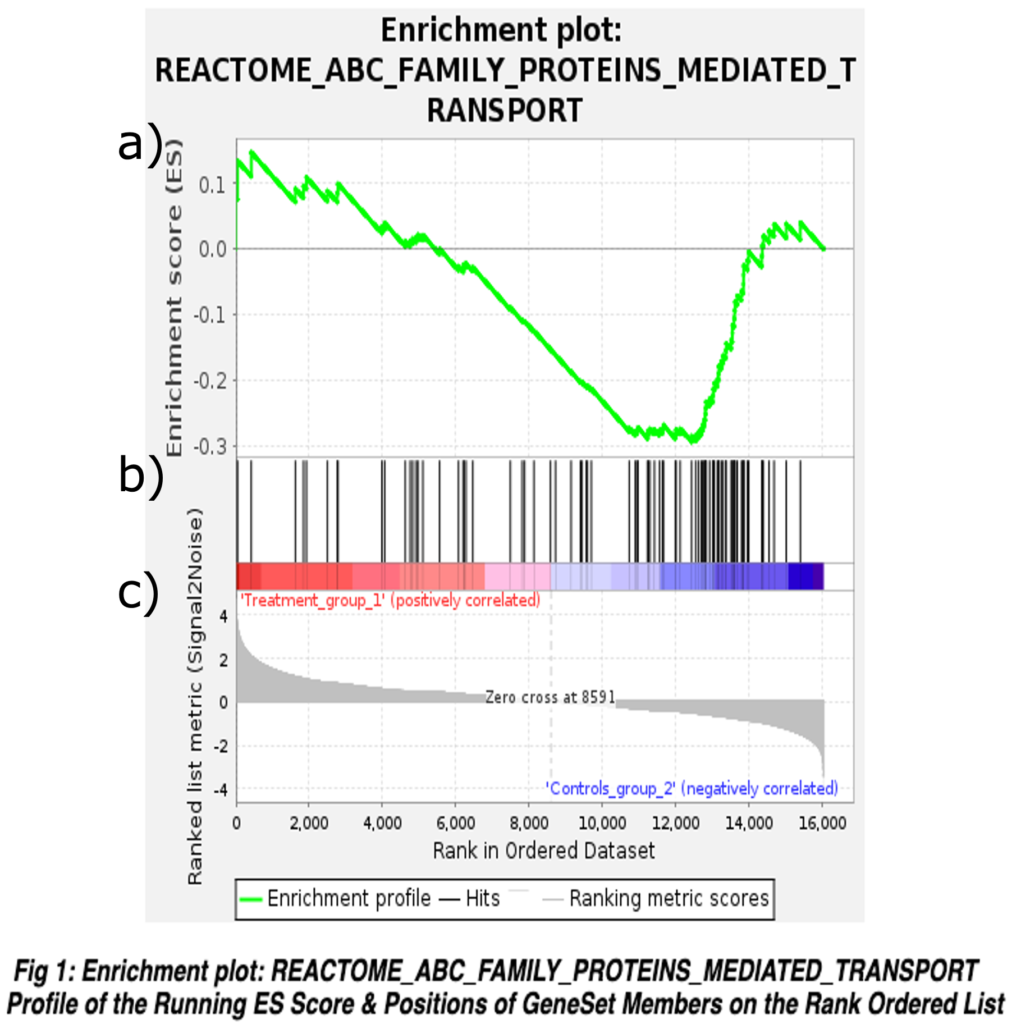
ES プロファイル
図8aでは、x軸はパスウェイにおける遺伝子のランクに対応します。一方、Y軸はESを表します。また、緑色の線はES曲線です。ここでは、リストを左から右に移動させたときのパスウェイのエンリッチメント度を表します。
この例では、0の水平線より上の値はポジティブのESです。これらの遺伝子はトリートメントグループと相関があります。一方、0 の水平線より下の値はネガティブの ES です。つまり、これらの遺伝子はコントロールグループと相関があります。
この例では、曲線は正のESから始まり、徐々に減少します。また、最低点はプロットの中央付近です。ES曲線がゼロから一番離れているポイントでESが計算されます。
遺伝子セットメンバーの位置
図8bでは、横軸に沿った黒い縦棒は、パスウェイの遺伝子がランク付けされたリストに現れる位置を表します。縦棒のほとんどが左側に集まっている場合は、ポジティブのエンリッチメント(トリートメントグループ方向)を示します。逆に、それらが右側に集まっている場合は、ネガティブのエンリッチメント(コントロールグループ方向)を示します。この場合、線は右側に集中しています。つまり、トリートメントグループ(赤斜線部分)に比べてコントロールグループ(青斜線部分)にネガティブのエンリッチメントがあることを示唆しています。
ランクリスト相関プロファイル
図8cはパスウェイの各遺伝子のランキングメトリックスコアです。ランキングメトリックでは、表現型と遺伝子の相関を測定します。この例では、評価指標は左側で高く正の値から始まり、右側に向かって減少します。また、遺伝子リストの特定のランクにゼロクロス点がマークされています。
パスウェイに関連する発現変動遺伝子のリスト
図9は、特定のパスウェイに関連する発見変動遺伝子のリストです。
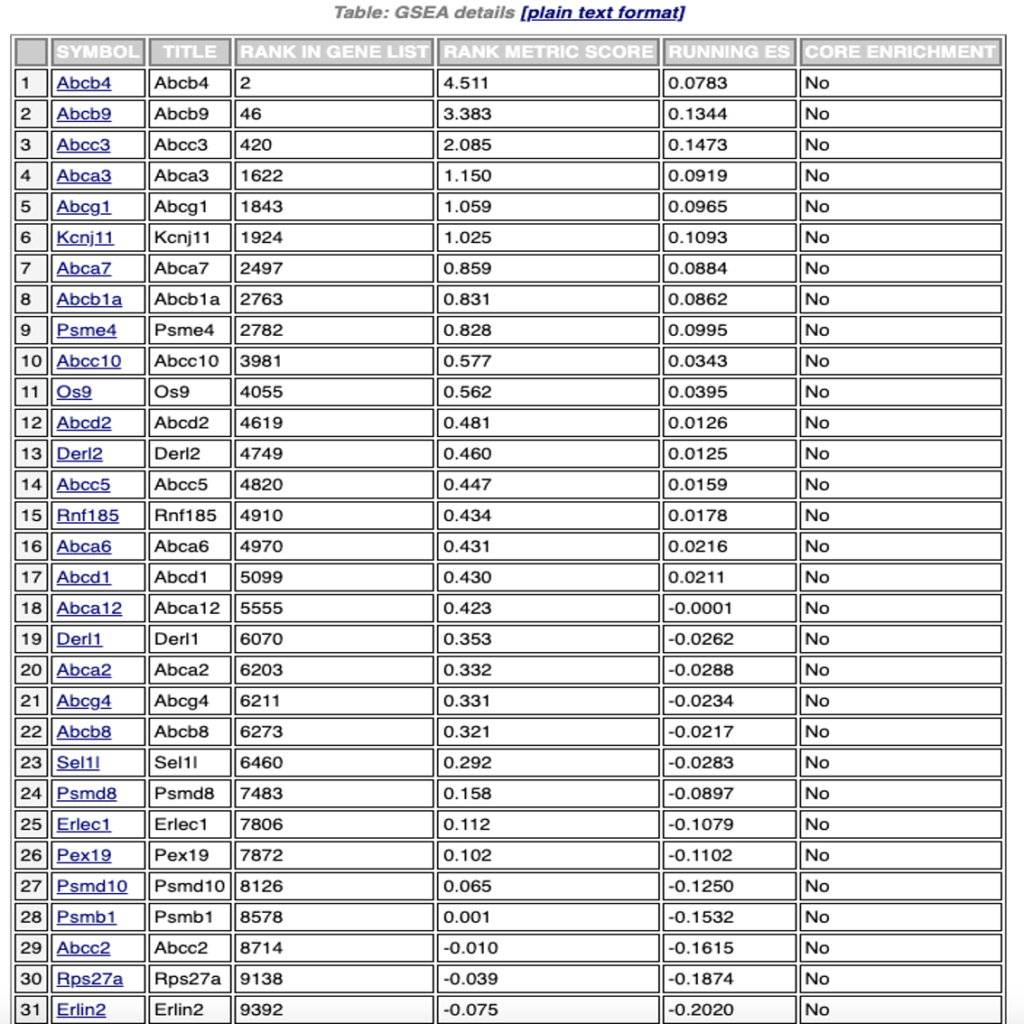
用語
| 用語 | 定義 |
| Symbol | 遺伝子の記号または識別子 |
| Title | 遺伝子の説明 |
| Rank in gene list | データセットから得られた遺伝子のランク付けされたリストにおけるその遺伝子の位置 |
| Rank metric score | 表現型と遺伝子の相関を表すスコア |
| Running ES | ランク付けされたリストの下に行くにつれてスコアを追跡する実行エンリッチメントスコア(ES) |
| Core enrichment | その遺伝子がコアエンリッチメントセットの一部であるかどうかの表示 |
パスウェイの遺伝子の発現レベルを示すヒートマップ
図10は特定のパスウェイに関連する遺伝子の発現レベルを示したヒートマップです。また、異なるサンプル間で遺伝子発現がどのように異なるかも示しています。

ここでは、各縦列は異なるサンプルを表します。一方、同じグループのサンプルは、同じ色で表されます。例えば、最初のサンプルセットはトリートメントグループ(オレンジ色)を表します。2番目のサンプルはコントロールグループ(グレー)を表します。各行はパスウェイの特定の遺伝子を表します。
この図では、配色は異なるサンプル間の各遺伝子の発現レベルを表します。赤はアップレギュレーションまたは高発現を表します。青はダウンレギュレーションまたは低発現を表します。一方、白は不変の発現を表します。
ランダムES分布プロット
図11は特定のパスウェイのランダムES分布プロットです。このプロットは、パスウェイの濃縮の有意性を評価するために使用されます。これは、観測されたESとランダムに並べ替えられたパスウェイからのESスコアの分布を比較することによって行われます。観測されたパスウェイのエンリッチメントが統計的に有意かを理解するのに役立ちます。
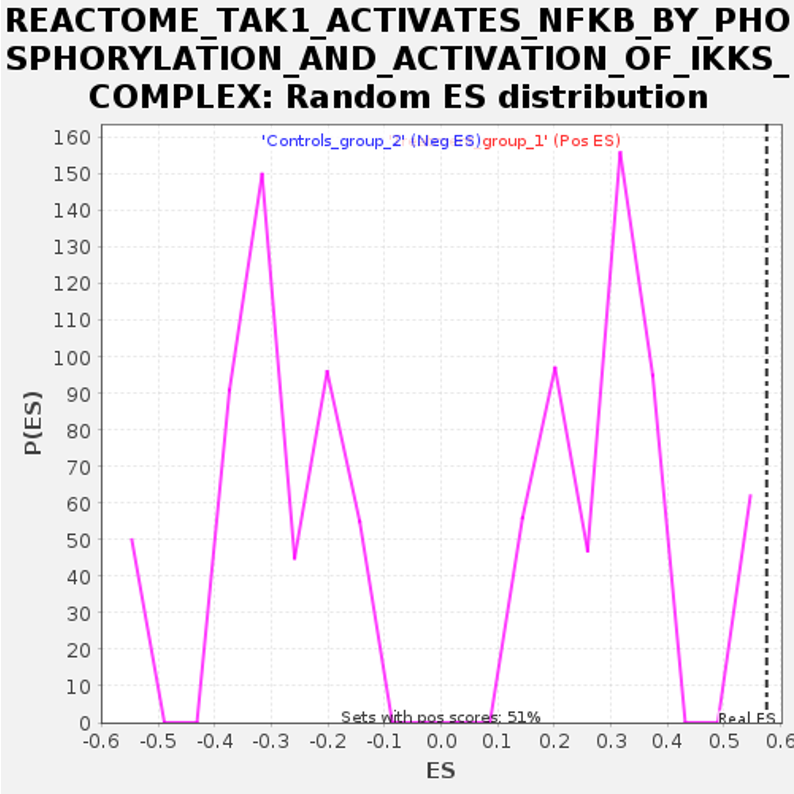
x軸とy軸
図11では、x軸はパスウェイをランダムに並べ替えて生成されたESを表しています。ESは、パスウェイに含まれる遺伝子が、ランク付けされた遺伝子リストの上位または下位にどれだけ強く過剰発現しているかを測定します。例えば、ES値がポジティブの場合は、トリートメントグループとのポジティブの相関を示します。一方、ネガティブの値はコントロールグループとのネガティブの相関を示唆します。
また、Y軸は遺伝子セットのランダム順列の確率密度関数P(ES)を示します。つまり、これは偶然に特定のESが得られる可能性を示しています。値が高いほど、そのESが順列中に現れる頻度が高いことを意味します。
全体として
ランダムなES分布はある地点で明確なピークを持ちます。つまり、これは並べ替えの間に、あるESスコア(ポジティブとネガティブの両方)が他のスコアよりも頻繁に出現することを示唆しています。これらのピークは、実際のデータで観察されたESが本当に有意かどうかを評価するのに役立ちます。
Guide to interpret results
「Enrichment in phenotype」セクションで、「Guide to」をクリックすると、MSigDBが提供するGSEAユーザーガイドが表示されます(1)。

「Dataset Details」 セクション
このセクションでは、発現データセットに関する情報を提供します。

情報
| 情報 | 例 |
| データセット中の遺伝子またはフィーチャーの数 | The dataset has 16027 features (genes) |
| データセットを簡素化した後のデータセット中の遺伝子数。例えば、転写産物を各遺伝子の遺伝子シンボルに簡素化します。これにより、同じ遺伝子の異なるアイソフォームの複数の発現値ではなく、遺伝子ごとに1つの発現値にフォーカスすることで解析が簡素化されます | No probe set => gene symbol collapsing was requested, so all 16027 features were used |
「Gene Set Details」セクション
このセクションでは、パスウェイについての情報を提供します。

情報
| 情報 | 例 |
| サイズにより解析から除外されたパスウェイの数。また、フィルターに使用されたパスウェイの最小値と最大値も示します | Gene set size filters (min=15, max=500) resulted in filtering out 720 / 1604 gene sets |
| 解析に使用されたパスウェイの数 | The remaining 884 gene sets were used in the analysis |
| 解析されたパスウェイのリスト | List of gene sets used and their sizes (restricted to features in the specified dataset) (Figure 8) |
List of gene sets used and their sizes
「Gene set details 」セクションでは、 「gene sets used and their sizes」(赤枠)をクリックすると、解析されたパスウェイのリストがtsv形式で表示されます(図12)。
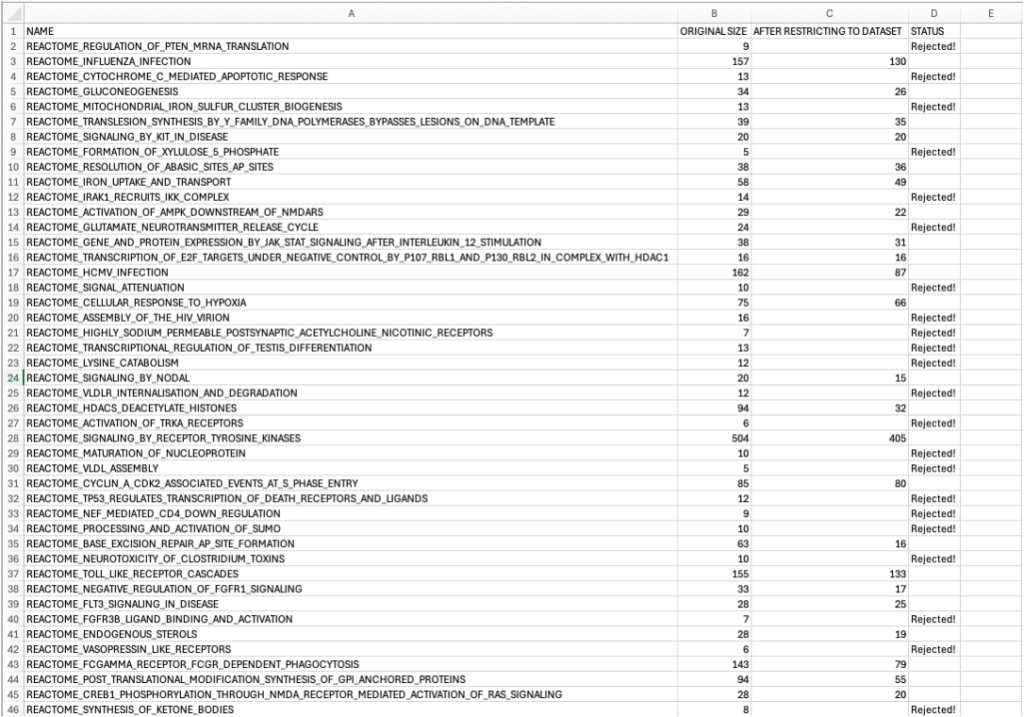
用語
| 用語 | 定義 |
| Name | パスウェイ名 |
| Original size | パスウェイに含まれる遺伝子の総数 |
| After restricting to dataset | インプットデータセットに実際に存在するパスウェイの遺伝子数 |
| Status | 解析におけるパスウェイのステータス。 空白:そのパスウェイが解析に使用しました Rejected:排除されたため解析に使用しませんでした |
「Gene Markers」セクション
このセクションでは、解析に使用した遺伝子のランク付けリストに関する情報を提供します。

情報
| 情報 | 例 |
| データセット中の遺伝子またはフィーチャーの数 | The dataset has 16027 features (genes) |
| データセット中の各表現型のマーカー数。すなわち、各表現型と相関のある遺伝子の数 | # of markers for phenotype Treatment_group_1: 8591 (53.6% ) with correlation area 57.4% |
| データセット中の遺伝子のランク順リスト(Excel形式)。各遺伝子の説明、遺伝子シンボル、遺伝子タイトル、ランクメトリックスコアなどの情報が含まれます | Detailed rank ordered gene list for all features in the dataset (Figure13) |
| 各表現型の上位50特徴のヒートマップ。ランク付けされた遺伝子と表現型の相関を示すプロットも示されています | Heat map and gene list correlation profile for all features in the dataset (Figure 14) |
| 遺伝子ランクとランキングメトリックスコアの正負の相関を示すバタフライプロット。デフォルトでは、バタフライプロットは上位100遺伝子を表示します。つまり、ランク付けされたリストの最初と最後の100遺伝子です | Butterfly plot of significant genes (Figure 15) |
Ranked order gene list
「Gene markers」 セクションで、「rank ordered gene list」(赤枠)をクリックすると、tsv形式のリストが表示されます(図13)。

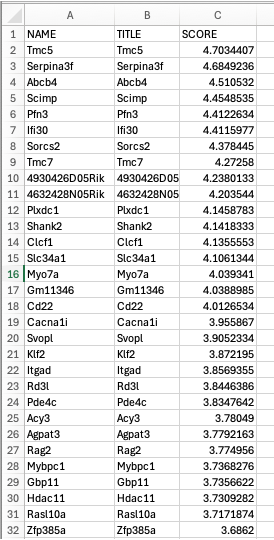
用語
| 用語 | 定義 |
| Name | ランク付けされたリストの遺伝子の遺伝子識別子 |
| Title | 遺伝子のフルネームまたは説明 |
| Score | ランク付けされたリストの遺伝子の順序付けに使用されるランク付け指標 |
Heat map and gene list correlation
「Gene markers」 セクションで 「heat map and gene list correlation」(赤枠)をクリックすると、各表現型にエンリッチされた上位50遺伝子のヒートマップをhtml形式で見ることができます(図14a)。また、ランク付けされた遺伝子リスト相関プロファイルも見ることができます(図14b)。

Butterfly plot
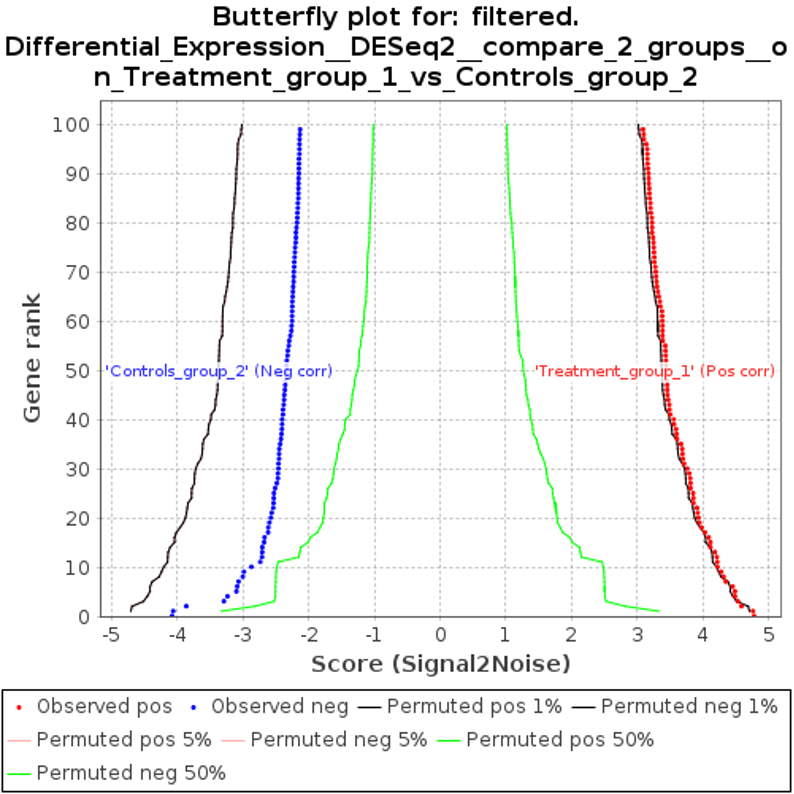
「Gene markers」セクションで 「butterfly plot」(赤枠)をクリックすると、異なるサンプルグループ間のバタフライプロットを見ることができます(図15)。このプロットは遺伝子スコアの順位分布を見るために使用します。

x軸とy軸
図15では、x軸はシグナル対ノイズ比を示しています。この比率は、各遺伝子が各サンプルグループとどの程度強く相関しているかを決定するためのスコアです。一方、Y軸は遺伝子のランクを表します。この順位は、x軸のスコアとの関連性によって並べられます。ランクが高いほど、サンプルグループと最もポジティブまたはネガティブの相関がある遺伝子と関連しています。
全体として
赤線は、トリートメントグループとポジティブの相関がある遺伝子を示します。青い線は、トリートメントグループとネガティブの相関がある遺伝子を示します。すなわち、コントロールグループと相関のある遺伝子です。
緑色の線は、グループ間に実際の差がなかった場合に予想される遺伝子の順位分布を示します。言い換えれば、ポジティブとネガティブ両方の場合の順列化されたランダム分布です。複数の緑の曲線は、1%、5%、50%といった閾値での無作為順列集合の異なるレベルを表します。
観測されたポジティブ(赤)とネガティブ(青)の曲線は、緑の順列化された曲線から逸脱しています。これは、実際のデータがランダムな分布とは大きく異なっていることを示しています。このことは、2つのグループ間で遺伝子発現に実際の差があることを示唆しています。
「Global Statistics and Plots」 セクション
このセクションでは、遺伝子セットとエンリッチメント結果に関する追加情報を提供します。
このセクションには以下が含まれます:
- 正規化ES(NES)とp値のプロット
- 遺伝子セット間のグローバルESヒストグラム
Plot of p-values vs. NES
「Global statistics and plots」セクションで、「p-values vs. NES」をクリックすると、p値とNESのプロットを見ることができます(図16)。

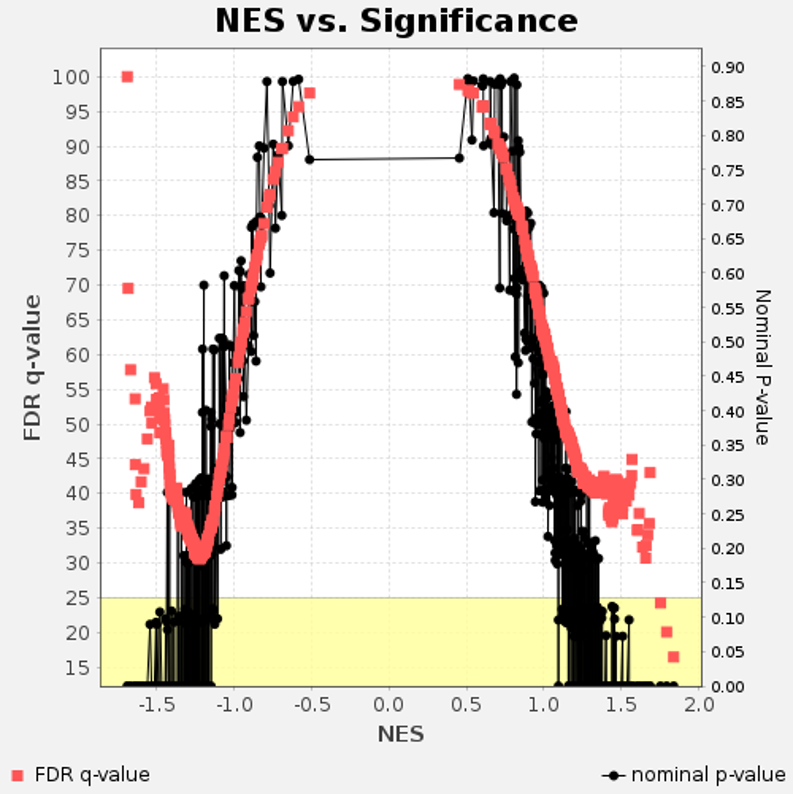
x軸
図16において、x軸は正規化エンリッチメントスコア(NES、normalized enrichment score)を表します。これは、遺伝子発現データにおいてパスウェイがどの程度エンリッチされているかを示す指標です。ポジティブのNES値は、パスウェイがトリートメントグループで発現増加またはエンリッチされていることを示唆します。ネガティブの NES 値は、コントロールグループでパスウェイがダウンレギュレートまたはエンリッチされていることを示唆します。
y軸
左のy軸はFalse Discovery Rate (FDR) q値を表し、補正された有意性の指標です。これは、有意な発見のうち偽陽性の割合を推定します。FDR値はプロットでは赤い四角で示されています。
右のy軸は名目上のp値(補正前のp値)を表します。名目上のp値は濃縮の統計的有意性を示します。p値が低いほど、観察されたエンリッチメントがランダムな偶然によるものではない可能性が高いことを示唆します。これらはプロット上の黒い三角形で表されています。
全体として
図16では、NES値が0より大きいパスウェイはトリートメントグループと関連しています。これらのパスウェイは、FDRと名目上のp値が低い傾向があります。これは有意なポジティブのエンリッチメントを示しています。一方、NESが0より小さいパスウェイ(左側)は、有意なネガティブのエンリッチメントを示します。これは、コントロールグループとの相関が高いことを示します。
Global ES histogram
「Global statistics and plots」セクションで 、「Global ES 」をクリックすると、遺伝子セット間のグローバルESヒストグラムを見ることができます(図17)。
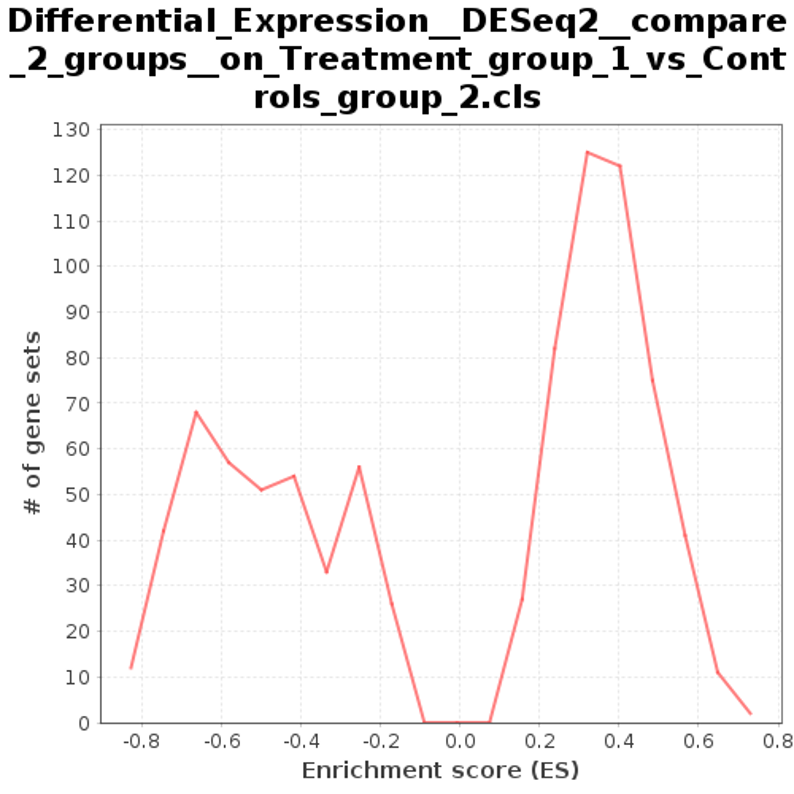
図17のx軸はESを表します。ESは、あるパスウェイが遺伝子のランク付けされたリストの上位または下位にどれだけ過剰発現しているかを示す尺度です。ポジティブのES値は、トリートメントグループでよりエンリッチされたパスウェイを表します。一方、ネガティブのES値は、コントロールグループでよりエンリッチされたパスウェイを示します。
Y軸は各ESに対応するパスウェイの数を表します。赤線は、ESに基づくパスウェイの分布を表します。
図17では、パスウェイの大半がポジティブのESを示します。これは、トリートメントグループで発現が増加していることを意味します。ネガティブのエンリッチメントを示すパスウェイは少ない(0の左側)です。これらのパスウェイは、おそらくトリートメントグループでダウンレギュレートされ、コントロールグループとの関連性が高いです。
参考文献
1. MSigDB. (2024). Gene set enrichment analysis (GSEA) user guide. MSigDB.
