すでにご存知かもしれませんが、BasepairはChIP-seq解析のための非常に人気のあるツールを多数提供しており、FASTQファイルをシームレスに有意義な結果へと導きます。約1時間で、トリミング、アライメントから、ピークコール、アノテーション、モチーフ濃縮、そして今回のパスウェイエンリッチメント解析までの全てステップを実行します。
BasepairのChIP-seqパイプラインの概要
Basepairでは、パイプラインで提供するツールを選択する際、最も信頼性が高く、引用回数の多いツールを選ぶように努めています。Basepairのバイオインフォマティシャンは常に最新の文献に目を通し、定期的にバージョンを更新することで、Basepairを利用する研究者が利用可能な最良のパイプラインを使用し、データから最良の結果を得られるようにしています。

BasepairのChIP-seqパイプラインで使用されるステップとツール。
BasepairのChIP-seqパイプラインで使用しているツールは以下の通りです。トリミングと品質管理はfastpを用いて行います。アライメントにはBowtie2を使用します。重複はSambambaを用いて除去します。ピークはMACS1.4またはMACS2を用いてコールされ、アノテーションされます。最後に、HOMERを用いてモチーフ濃縮とパスウェイ濃縮解析を行います。
ChIP-seqデータのパスウェイエンリッチメント解析
Basepairは濃縮解析を行うためにHOMERのfindGOツールを使用しています。これはGene OntologyやMSigDBなどの一般的なデータベースから20以上のオントロジーと遺伝子セットを用いて濃縮度をチェックする非常に人気のあるツールです。
データはインタラクティブな表に表示されます。データベースカラムの横にあるフィルターアイコンをクリックすると、特定のデータベースのパスウェイをフィルターして表示することができます。
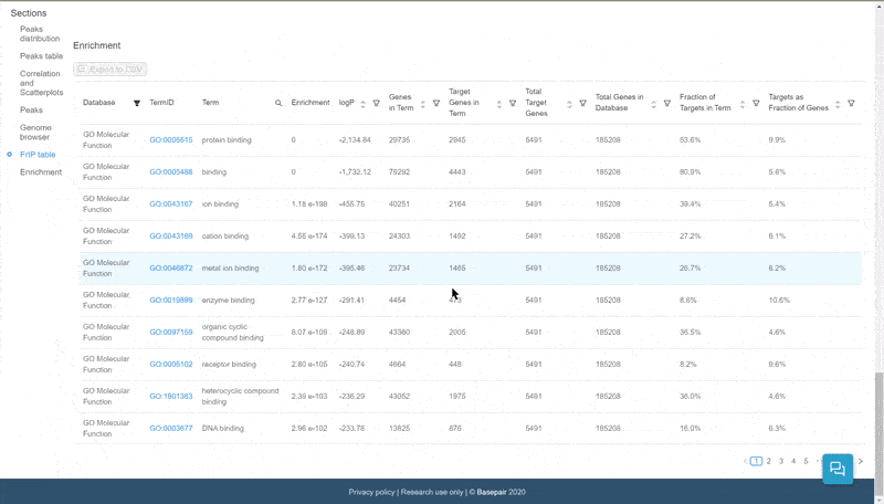
12以上のデータベースから経路を素早く選択し、フィルタリング。
ChIP-seqレポートのその他のセクション
エンリッチメントの表に加え、BasepairのChIP-seqレポートには他にもインタラクティブな図表が多数掲載されています。
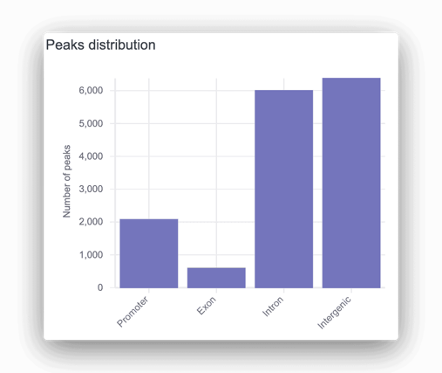
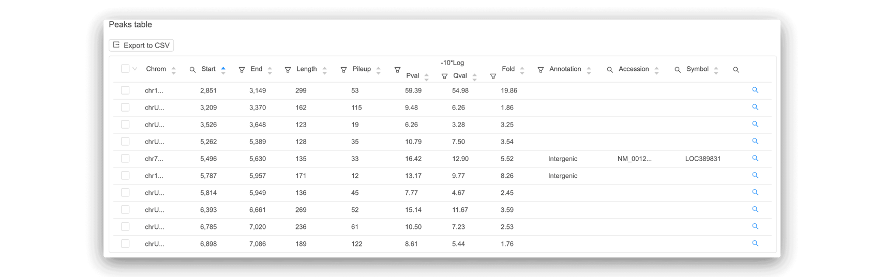
ピーク分布グラフとピークコールテーブルは、データ中のシグナルピークを簡単に特定するのに便利です。各ピークはプロモーター、イントロン、遺伝子間としてアノテーションされ、対応する遺伝子が表示されます。検出されたピークについて、モチーフ解析が行われ、転写因子結合部位が過剰に存在することがわかります。
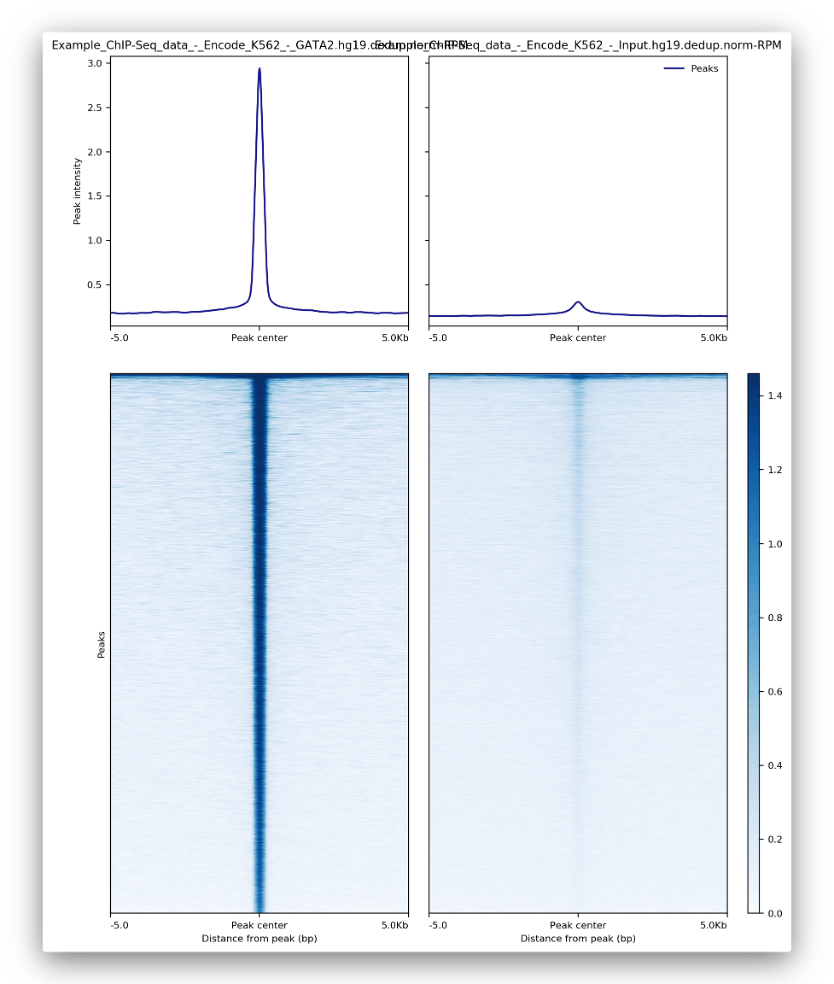
遺伝子本体と転写開始部位の図と対応するヒートマップは、適切なシグナルを見つけるのに役立ちます。これにより、サンプルは選択したChIPに期待されるパターンを確実に示します。シグナル対ノイズ比が低い場合、これらの図は問題を素早く特定するのに役立ちます。
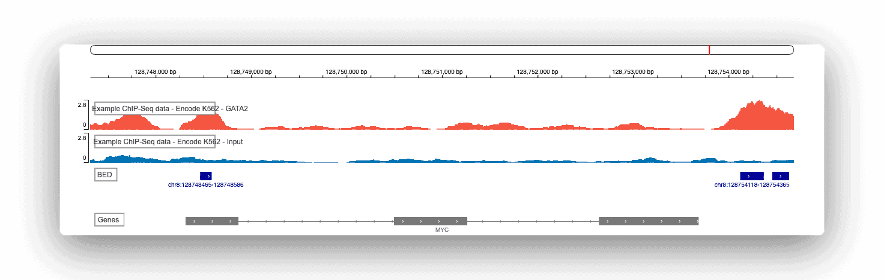
ゲノムブラウザは生のゲノムデータの概要を視覚的に確認できる重要なツールで、すべてのChIP-seqレポートの最後に含まれています。ズームイン、ズームアウト、特定の遺伝子や遺伝子座の検索が可能です。
今すぐChIP-Seqデータを解析して、30分で結果
BasepairのChIP-seqパイプラインは、平均するとわずか30分で完了します。すでに米国の一流大学や病院の何百人もの研究者が、エピジェネティック研究のスピードアップと結果の迅速な発表のためにBasepairを利用しています。
すでにChIP-seqデータをお持ちの方で、このパイプラインを試してみたい方は、無料トライアルにご登録ください。6サンプル分のフリートライアルを進呈しています。サンプルごとの無制限解析と12ヶ月の保存を含む追加サンプルは期間限定でそれぞれわずか6,000円(税別 2025年4月現在)です。
この記事は「Pathway Enrichment Analysis for ChIP-seq Data」を翻訳、再構成したものです。
