Basepairは、fastqなど生データがあれば、NGSデータの解析ができます。一方で、自分で解析することが重要・必要なケースも少なからずあると思います。とはいえ、自分でやるべきか、判断に迷うこともあると思います。そこで、コストパフォーマンスとタイムパフォーマンス(コスパとタイパ)は、どのくらいになるか検討してみたいと思います。
検討方法
「次世代シーケンサーDRY解析教本(改訂第2版)」を参考に以下の内容で比較しました。
解析項目
- DEG(Differential Expression Genes)解析。
サンプルデータ
SRAで公開されている、SRP04500のデータを使用。
- 抽出条件
- cell type:cd4
- diseasestatus:type1 diabetes、healthy control
- gender:female
- 使用データ(アクセッションナンバー)
- GSM1479441
- GSM1479457
- GSM1479463
- GSM1479502
- GSM1479509
- GSM1479523
- GSM1479543
比較方法
- 自分で解析する場合:「次世代シーケンサーDRY解析教本(改訂第2版)」の情報
- Basepairを使う場合:実際にデータをダウンロードし、Basepairで解析
自分で解析する場合
自分で解析する場合は、マシンの用意、ソフトウェアの整備などが必要です。
マシンの選定
参考図書のおすすめはMacなので、Macで考えることにします。たとえばSTARでは、32GBのメモリが必要とされています。また、ひとまず1TBのストレージをつけておきます。経済合理的に思える範囲で検討すると、以下のような構成が考えられます。その他にも、外付けHDD4TB(1万円程度)などアクセサリーが必要になります。ちなみに、AppleのRetinaディスプレイが視認性が良いため個人的には好みですが、それだけで30万くらいします。
| Mac Miniの場合 | MacBook Pro 14inchの場合 |
|---|---|
| 10コアCPU、16コアGPU、16コアNeural Engine搭載Apple M2 Proチップ 32GBユニファイドメモリ 1TB SSDストレージ | 12コアCPU、18コアGPU、16コアNeural Engine搭載Apple M3 Proチップ 36GBユニファイドメモリ 1TB SSDストレージ |
| 274,800円(税込) キーボード、マウス、ディスプレイは別途 | 458,800円(税込) |
セットアップ
買ってきたMacは、そのままでは使えません。バイオインフォマティクスに使えるようにするためにはセットアップが必要です。たとえば、以下のようなものがあります。各解析を行うためには、それぞれ必要なプログラムをインストールする必要があります。
- R:統計やグラフを描く
- Homebrew:パッケージ管理プログラム
- wget:ダウンロードするためのプログラム
- Bioconda:Pythonのパッケージ
- SRA Toolkit:SRAが提供するツール
ちなみに、これらの作業はR以外は、コマンドラインで行います。
解析する
まず必要なのは、サンプルデータとレファレンスデータです。今回サンプルデータは、SRAから入手したデータを使いますが、レファレンスデータは、対象のゲノム配列が記述されているレファレンスシーケンスと、遺伝子などのゲノム上の位置情報を記述したアノテーションファイルを別途用意する必要があります。次に、STAR、RSEM、DESeq2などを順次使います。
所要時間
参考文献の「再現・検証」にある日数・時間を抜き出してみます。
解析前のもろもろセットアップは完了していたようで、省略されています。その後工程を見ると、作業を行った日数は9日、作業時間は13時間40分。また、8サンプルの読み込みと圧縮ファイル作成が完了したのが、約6日後とあります。また、作業全体としてはトータル30日かかったようです。開始日が5月19日、最終日が6月18日とあります。
Basepairを使う場合
参考文献の「Level2:実践編にRNA-Seq」の記述に沿って、これと同様の内容をBasepairでやってみます。完全に一致はしていませんが、概ねやりたい内容としては同等であろう、ということでご容赦ください。
セットアップ
セットアップはアカウントを作成するだけです。マシンのセットアップなどは不要です。コストは、7サンプルので6,000×7=42,000円。税込だと、46,200円となります。(価格表参照)
GEOからのデータのダウンロード
Basepairには、”Import data from GEO”という解析項目があります。これは、アクセッションNo.を指定すると、該当するfastqを探し出しダウンロードしてくれます。120分かかりました。ちなみに、Basepairにはレファレンスゲノムはあらかじめ用意されていますので、あらためて用意する必要はありません。
解析する
Basepairには、DESeq2が用意されています。このDESeq2は、名前はDESeq2ですが、fastqを与えると、先にSTARを自動的に走らせてくれます。なので、ファイルを指定して、DESeq2を選ぶだけで完了です。(詳しくはRNA-Seqもご覧ください。)
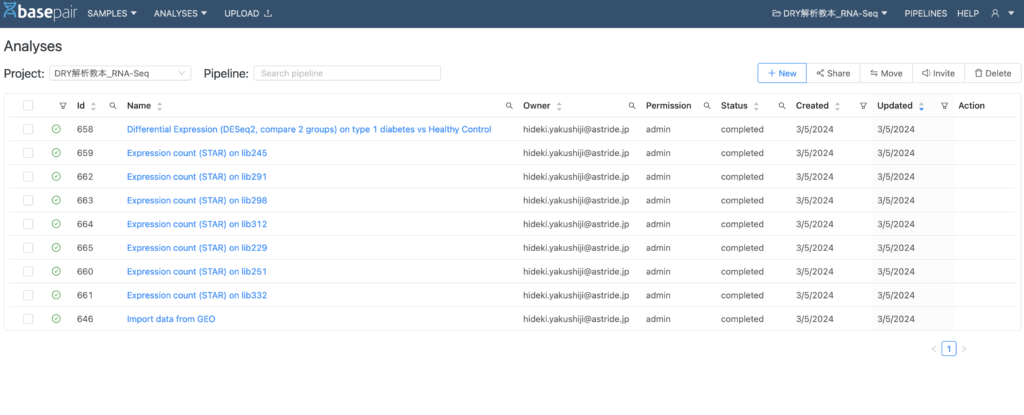
下の図は、id:685 Differential Expressionを走らせた結果です。その下の659、662など、それぞれのサンプルに対してSTARが走ったことがわかります。Differential Expressionのidが、一番若いことでもそれがわかります。
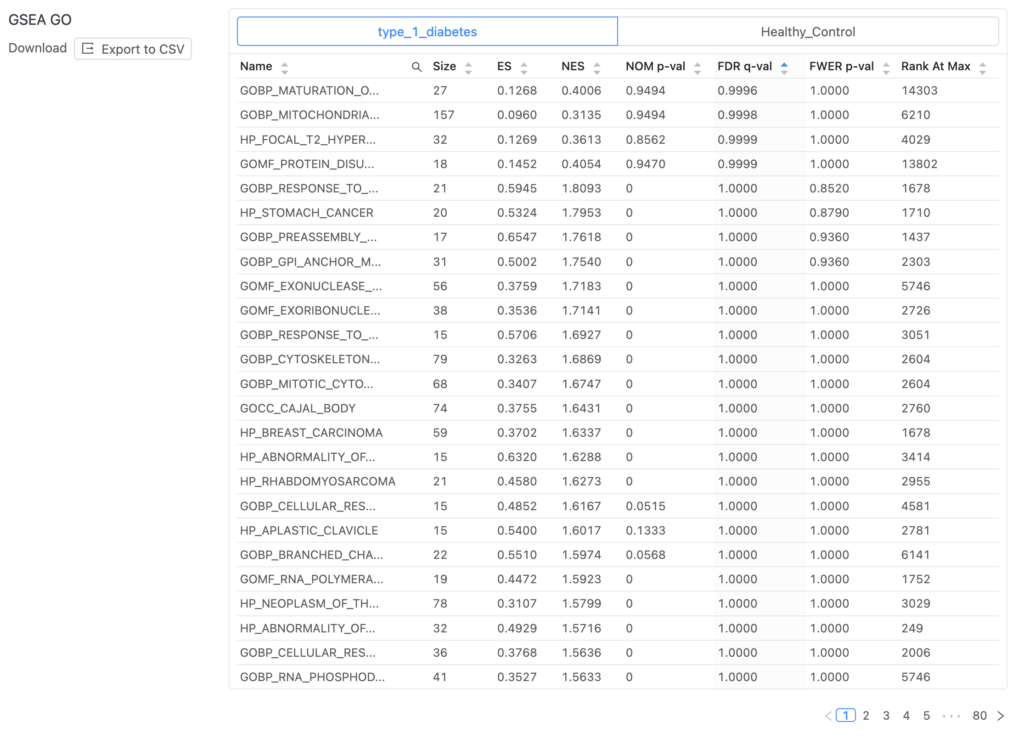
また、BasepairのDifferential Expressionは、GSEAも行ってくれます。GOとPathwayのテーブルが生成されます。グルーピングした通り、Type_1_diabatesとhealth_controlのタブがあります。

所要時間
GEOからのダウンロードが2時間(120分)、それぞれのSTARに平均24.8分(20〜29分)、DESeq2に27分かかっていました。画面の左上に所要時間が表示されています。Basepairは、解析を同時並行に行うことができるので、STARの所要時間は、24.8×7ではなく、30分程度ということになります。
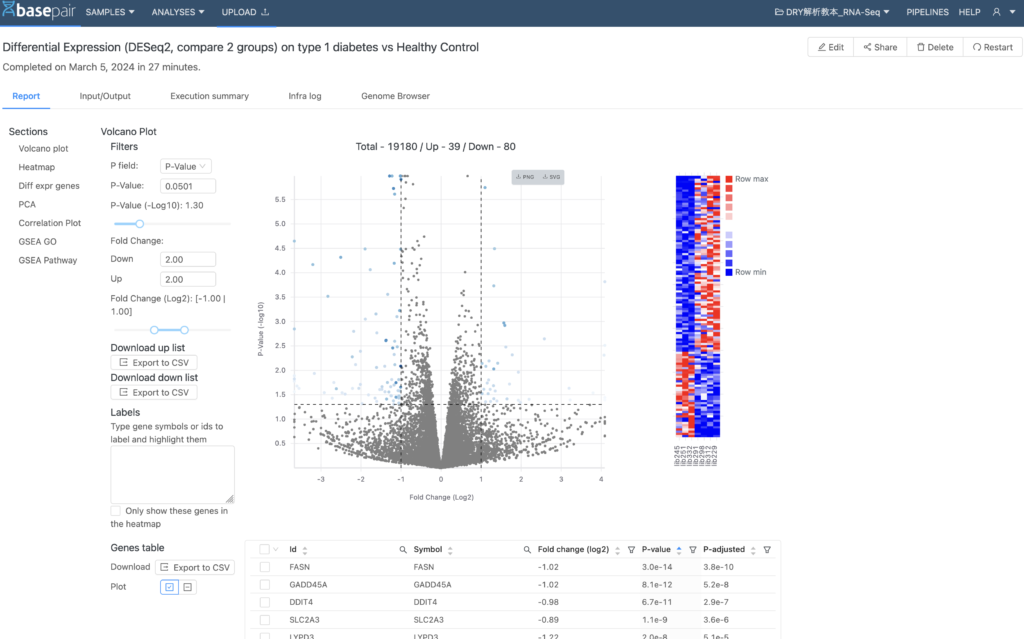
ダウンロードからDESeq2が完了するまで、その日のうちに終えることができました(上の解析リストの表示が同じ日になっているのに注目してください)。ちなみに、実作業時間はGEOのファイルを選ぶのが一番手間取って1時間くらいかかりましたが、それ以降はBasepair上の作業で5分もかかりませんでした。
比較
表にまとめると、このようになりました。コスト、所要時間もBasepairの方が安く、短くなりました。「自分で解析する」方は、トータルで30日かかっています。一方で、Basepairでは1日で完了しました。
| 自分で解析する | Basepairを使う | |
|---|---|---|
| コスト(税込) | 278,000円〜 | 46,200円 |
| 所要時間 | 作業日数:9日 作業時間:13時間40分 サンプルファイル準備:約6日 | ダウンロード:120分 STAR:平均24.8分 DESeq2:27分 実作業時間:1時間 |
| 完了までの日数 | 30日 | 1日 |
考察
所要時間
「自分で解析する」方は、トータルで30日かかっています。もちろん、他の仕事の傍らで確保できる時間で進められたのだと思います。とはいえ、研究者もほかにやらなければならないことはたくさんあります。ということ考えると、状況としてはさほど違いはないと思われます。また、ファイルの準備でかかっている6日の間は何もできないということになります。Basepairを使う場合は、すぐに始められ、解析も迅速に行えます。
コスト
この程度サンプル数では、コスト面ではBasepairに軍配が上がります。ですが、サンプル数が多くなればなるほど、自分で解析する方が安くなるでしょう。マシンは一定程度の期間で買い替えることになります。なので、サンプルあたりのマシンコストを計算してみるのも一考かもしれません。
マシン負荷
サンプル数多い場合は、自分で解析する方が良さそうです。ただし、同時に解析の所要時間を考える必要があります。多くの場合、自分で用意したマシンではリソースに限りがあります。モンスターマシンでない限り、解析を並列に実行することは難しいと思われます。つまり、サンプル数の分だけ時間を要することになります。また、計算中はCPU、メモリを一杯一杯使うことになりますので、他の計算はできないと思っておいた方がよいでしょう。
一方で、Basepairは並列実行ができるため、サンプル数によって所要時間に差がでません。また、クラウド上で計算が実行されるので、自分のマシンの負荷はありません。
まとめ
バイオインフォマティクスを自分で行うのは、非常に有益です。解析のロジックやアルゴリズムを理解していることで、より深い洞察が得られるということも重要な点です。とはいえ、サンプル数が多い・少ないにかかわらず、コスト・所要時間を考えるとのBasepairのようなクラウドツールを使うのも選択肢の一つとして検討の価値はあるでしょう。
今回行った解析の結果をご覧になりたい方はお問い合わせからご連絡ください。そのままシェアします。
最大6サンプル フリートライアル 実施中
最大6つのサンプルを無料でアップロードして分析できます。アップロードされたサンプルに対する解析は無制限です。世界トップクラスの機関、研究室、製薬チームがBasepairを使用して、数千ドルを節約している理由をご覧ください。

