はじめに
モチーフ検出とは、繰り返し現れる短いDNA配列を検出することです。エピジェネティクスでは、転写因子や他の制御タンパク質の結合部位を表します。
モチーフの検出はピークコーリングの後に行われます(図1)。ピークコーリングはピークを同定するプロセスです。ピークは転写因子などのタンパク質がDNAと結合しているゲノムの領域です。モチーフ検出では、ピーク内で過剰に発現している配列を同定します。
アノテーションは、検出されたモチーフを既知のモチーフに結びつけるプロセスです。これは同定されたモチーフをデータベースにあるものと比較することで行われます。これにより、モチーフの潜在的な生物学的役割を決定することができます。

図1. エピジェネティックデータのモチーフ発見とアノテーションを行うBasepairのパイプライン。* ATAC-Seq、ChIP-Seq、CUT&RUNとCUT&Tag。
パイプライン
モチーフ検出とアノテーションに使用されるパイプラインです:
- ATAC-Seq Peaks, Motif (MACS2)
- ChIP-Seq Peaks, Motif (MACS v2, Homer)
- CUT&RUN Peaks, Motif (MACS2, Homer)
- CUT&Tag Peaks, Motif (MACS v2, Homer)
結果 (「Report」タブ)
結果の場所と形式は、上記のすべてのパイプラインで同じです。ここでは、ATAC-Seqパイプラインの結果を例に説明します。「Report」タブ(赤枠)の「Motifs」セクション(青枠)にあります。
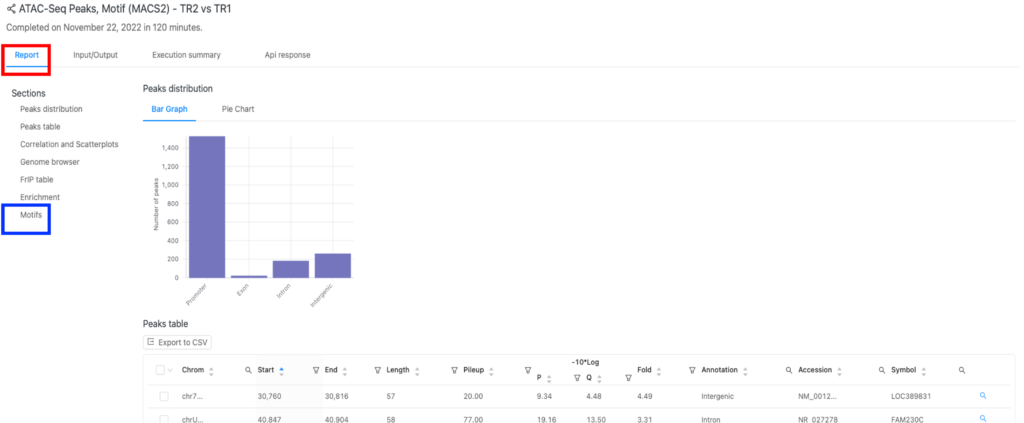
モチーフ発見とアノテーション
ピークで同定され、エンリッチされたモチーフの要約と、それらにマッチする転写因子結合プロファイルを図3に示します。
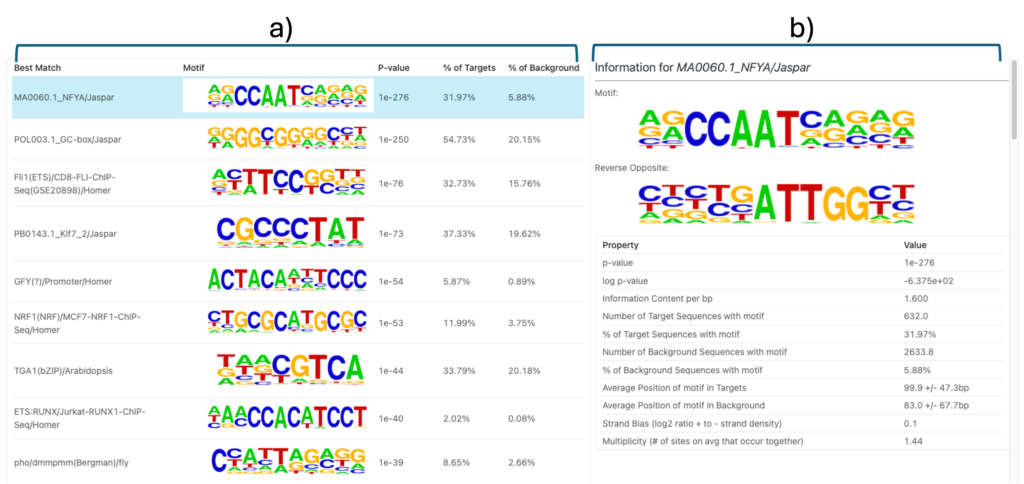
用語
| 用語 | 定義 |
| Best Match | 発見されたモチーフにデータベースから最も類似した既知のモチーフまたは転写因子結合プロファイル |
| Motif | 発見されたモチーフの配列ロゴ。背の高い文字は、その位置のヌクレオチドの保存性が高いことを示します |
| p-value | モチーフエンリッチメントの統計的有意性 |
| % of Targets | モチーフを含むピーク領域(ターゲット)の割合 |
| % of Background | モチーフを含むバックグラウンド領域(非ピーク領域)の割合 |
特定モチーフのアノテーション
図3aの特定のモチーフをクリックすると、図3bに詳細な要約が表示されます。
用語
| 用語 | 定義 |
| Motif | モチーフの配列ロゴ |
| Reverse Opposite | モチーフの配列ロゴの逆相補体 |
「Properties」用語
| 用語 | 定義 |
| p-value | エンリッチメントの有意水準 |
| log p-value | エンリッチメントの対数スケール有意性(p値) |
| Information Content per bp | モチーフの特異性と、モチーフの各位置で各ヌクレオチドがどの程度強く保存されているかの尺度 |
| Number of Target Sequences with motif | モチーフを含む標的配列の数 |
| % of Target Sequences with motif | モチーフを含むターゲットの割合 |
| Number of Background Sequences with motif | モチーフを含む背景配列の数 |
| % of Background Sequences with motif | モチーフを含む背景配列の割合 |
| Average Position of motif in Targets | 発見されたモチーフの標的配列内の平均位置 |
| Average Position of motif in Background | バックグラウンド配列中のモチーフの平均位置。バックグラウンド配列は、転写因子が結合しないと予想される領域 |
| Strand Bias (log2 ratio + to – strand density) | モチーフがDNAの順鎖(+)と逆鎖(-)のどちらに多く見られるかを測定します |
| Multiplicity (# of sites on average that occur together) | 一つの標的配列または領域内でのモチーフの平均出現数 |
モチーフ検出とアノテーションのファイル
図3bをさらに下にスクロールすると、「Files 」セクションがあります(図4)。

ファイルは以下の通りです。
| セクション | ファイルの説明 |
| Motif File | |
| -File (matrix) | 鎖上のモチーフのPWM(Position Weight Matrix)を含みます。PWMは、モチーフの各位置に各ヌクレオチド(A、C、G、T)が出現する確率を表します |
| -Reverse opposite | 反対(-)鎖上のモチーフを表すモチーフの逆相補体を含みます |
| PDF Format Logos | |
| – Forward logo | DNAの+鎖のモチーフ配列のグラフ表示(PDF形式) |
| – Reverse opposite | 鎖上のモチーフの逆相補配列のグラフ表示 |
| Peaks with Motif | |
| – Peaks | モチーフが同定されたピークのリスト |
| – Fasta | モチーフが見つかった領域の配列をFASTA形式で含みます |
| – Bed | モチーフが見つかったゲノム領域(ピークなど)をBED形式で含みます |
| Peaks without Motif | |
| – Fasta | モチーフが見つからないピークの配列をFASTA形式で含みます |
| – Bed | モチーフが見つからないピークのゲノム座標をBEDフォーマットで含みます |
ヒートマップ
図3bをさらに下にスクロールすると、ヒートマップが表示されます。これは異なるサンプルのピーク間のシグナル強度を比較したものです(図5)。
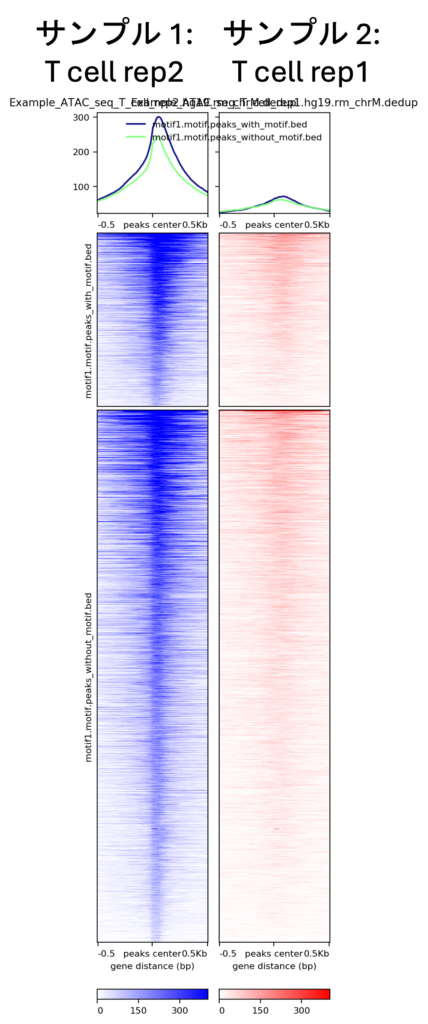
図5上部の折れ線プロットでは、x軸はピーク中心(0)からの距離を表しています。Kbから-0.5Kbまでの距離です。Y軸はシグナル強度を表し、これはこれらの領域にマップされたリードの数です。青い線はモチーフを含むピークのシグナルです。緑の線はモチーフを含まないピークのシグナルを表します。両サンプルの青い線は、緑の線に比べてピーク中心で強いシグナルを示しています。したがって、モチーフはクロマチンアクセシビリティの高い領域を表します。
図5では、上のヒートマップは選択したモチーフを含むピークのシグナル強度を示しています。下のヒートマップは上のヒートマップと似ています。ただし、選択したモチーフを含まないピークに焦点を当てています。X軸はピーク中心からの距離を示します。Y軸は個々のピークに該当します。下部のカラースケールは信号強度を示し、色が濃いほど強度が高いことを示します。
両サンプルとも、選択したモチーフを含むピークは、ピーク中心でより高いシグナル強度を示しています。これは、これらの領域においてクロマチンアクセシビリティが高いことを示しています。
既知のモチーフとのマッチ
図3bをさらに下にスクロールすると、選択されたモチーフと既知のモチーフの一致情報があります。既知のNFYA転写因子結合部位との一致の例を図6に示します。
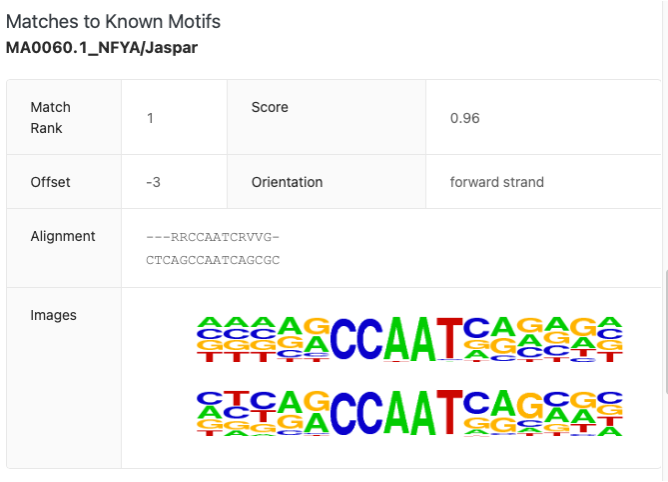
図6. 選択したモチーフと既知のNFYA転写因子結合部位との一致。
用語
| 用語 | 定義 |
| MA0060.1_NFYA/Jaspar | 選択された発見モチーフと一致する既知のモチーフまたは転写因子結合プロファイルの名前(例:JASPARデータベースで定義されたNFYA転写因子) |
| Match Rank | 選択された発見されたモチーフと既知のモチーフの間のマッチングのランクレベル。ランクは0から1までです |
| Score | 選択された発見されたモチーフと既知のモチーフの一致の強さまたは確信度。スコアは0から1の間で、スコアが「1」に近いほど、選択されたモチーフと既知のモチーフの類似性が高いです |
| Offset | 選択したモチーフと既知の転写因子とのアラインメント。数字は塩基数を表します。または+はゲノムの左右への塩基のずれを表します。 |
| Orientation | 選択したモチーフ上の既知の転写因子結合部位の位置(例:順鎖または逆鎖) |
| Alignment | 選択されたモチーフと既知のモチーフのアラインメント |
| Images | 既知のモチーフ(フォワード鎖とリバース鎖)のグラフ表示 |
図6では、選択したモチーフはNFYA転写因子結合モチーフと非常に強くマッチし、マッチランクは1、高信頼性スコアは0.96です。マッチは中央で揃わず、左に3塩基分シフトしています(offset -3)。モチーフは前方鎖(前方鎖の向き)に位置しています。
関連ブログ
- ATAC-seq解析 https://basepairtech.jp/analysis/atac-seq/
- ChIP-seq解析 https://basepairtech.jp/analysis/chip-seq/
- Cut&Run and Cut&Tag解析 https://basepairtech.jp/analysis/cutnrun-cutntag/
最大6サンプル フリートライアル 実施中
最大6つのサンプルを無料でアップロードして分析できます。アップロードされたサンプルに対する解析は無制限です。世界トップクラスの機関、研究室、製薬チームがBasepairを使用して、数千ドルを節約している理由をご覧ください。
