「DESeq2」パイプラインによって生成された全てのアウトプットファイルは「Input/output」タブ(赤枠 1)にあります。Reactome のパスウェイエンリッチメント結果は、「pathway.gsea.zip」フォルダ(青枠)にあります。これらのファイルは、論文発表や下流の解析に役立ちます。
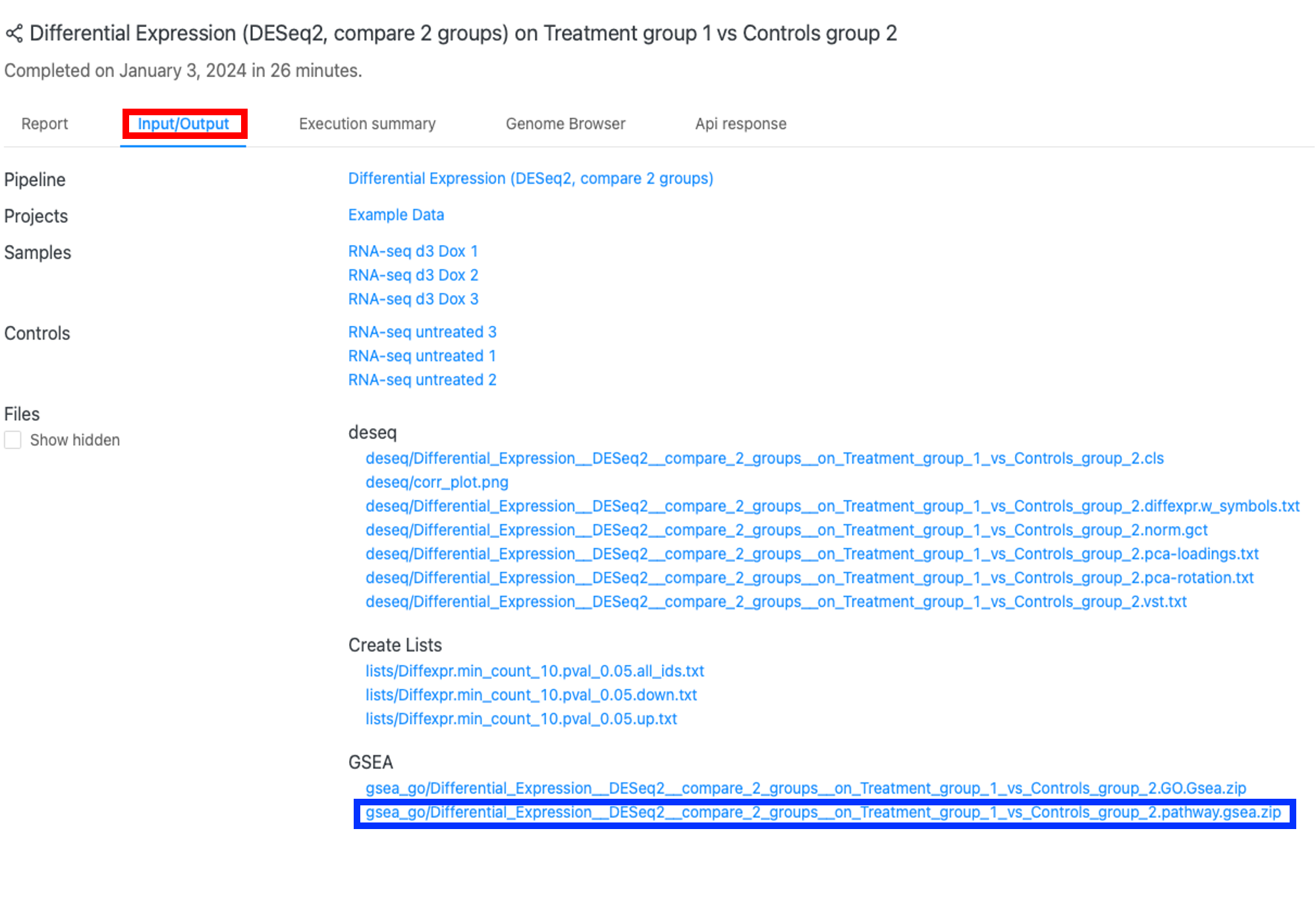
「pathway.gsea.zip」フォルダ内のファイル
GSEAレポート(index.html)
図2では、DESeq2によって生成されたGSEAレポートを示します。フィルタリング後の発現変動遺伝子リストに対して実行されたパスウェイエンリッチメント解析の結果が含まれています。

レポートの詳細はテクニカルブログ「Reactomeパスウェイエンリッチメント解析からアウトプットされたGSEAレポート」をご覧ください。
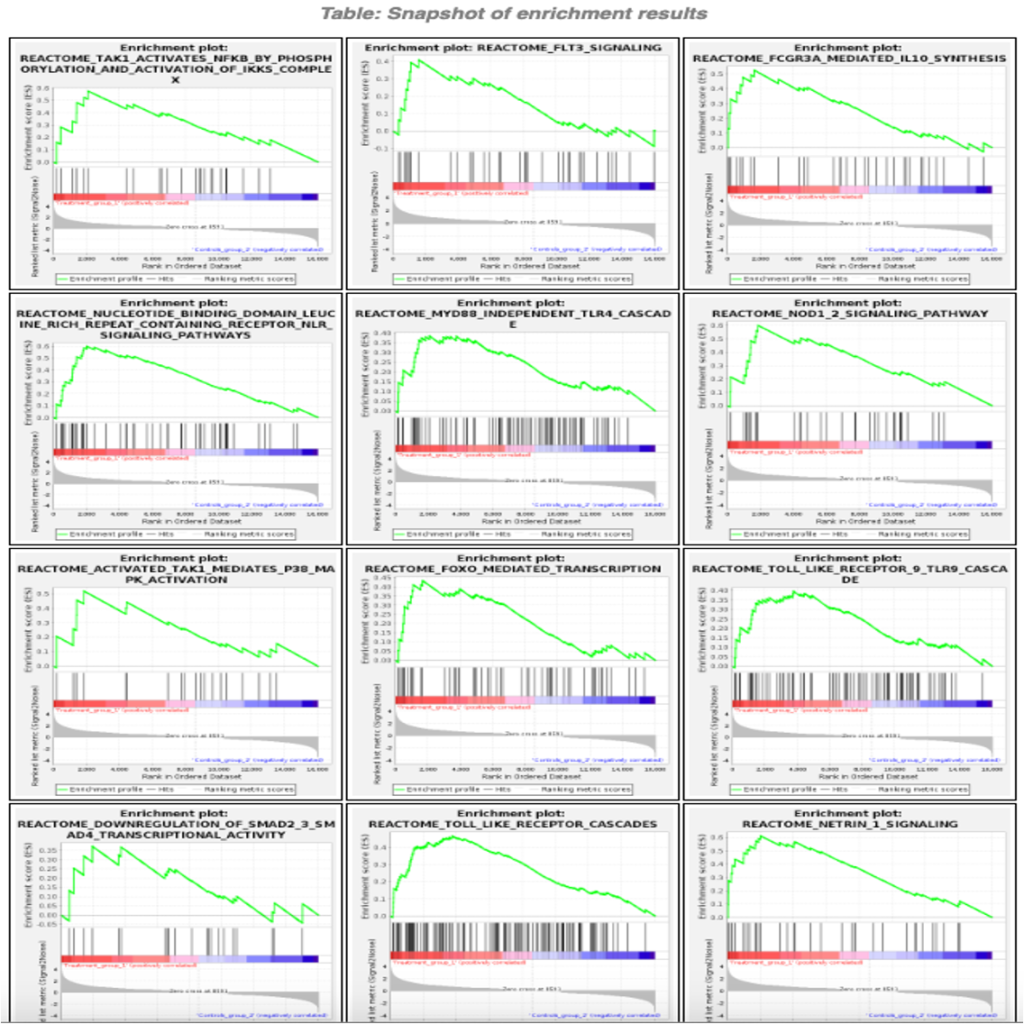
結果のスナップショット(pos_snapshot.html・neg_snapshot.html
図3では、陽性および陰性エンリッチメント結果のスナップショットを示します。
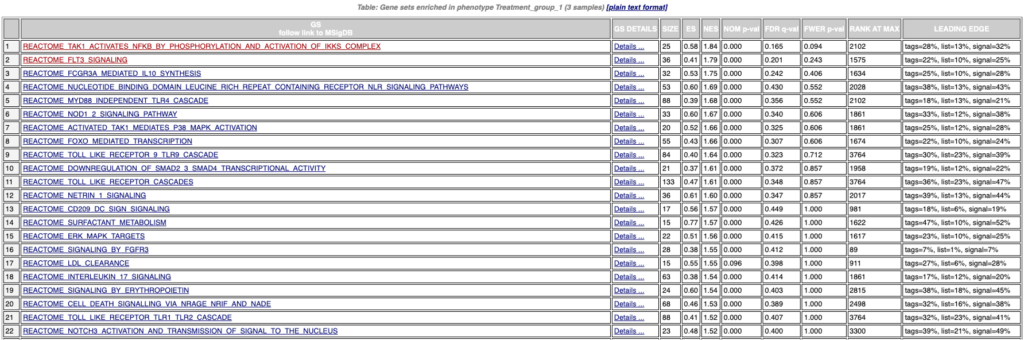
サンプルグループ内のエンリッチされたパスウェイのリスト(gsea_report_for_<SAMPLE>.html)
図4では発現変動データに基づいて、サンプルグループでエンリッチされたパスウェイのリストを示します。サンプルグループで、別のサンプルグループと比較して、特定のパスウェイがアップレギュレートまたはダウンレギュレートされているかどうかの情報を提供します(1)。
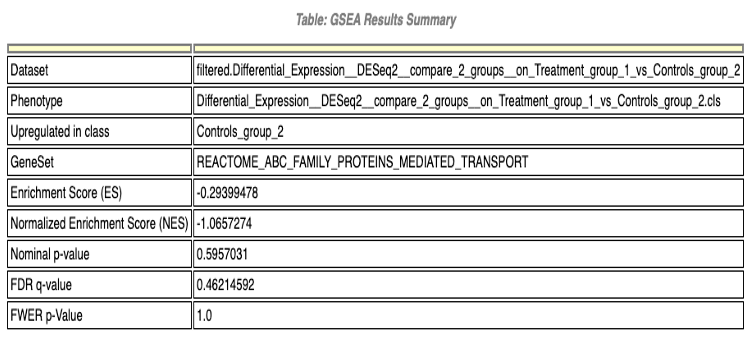
パスウェイの詳細情報(<pathway>.html)
図5〜9では、データセット中の発現変動遺伝子の中で有意にエンリッチされた特定のパスウェイの詳細情報を示します。内容は以下の通りです:
- パスウェイエンリッチメント統計
- パスウェイのエンリッチメントプロット
- パスウェイに関連する発現変動遺伝子のリスト
- パスウェイにおける遺伝子の発現レベルを示すヒートマップ- ランダムエンリッチメントスコア分布プロット
enplot_REACTOME_<pathway>.png
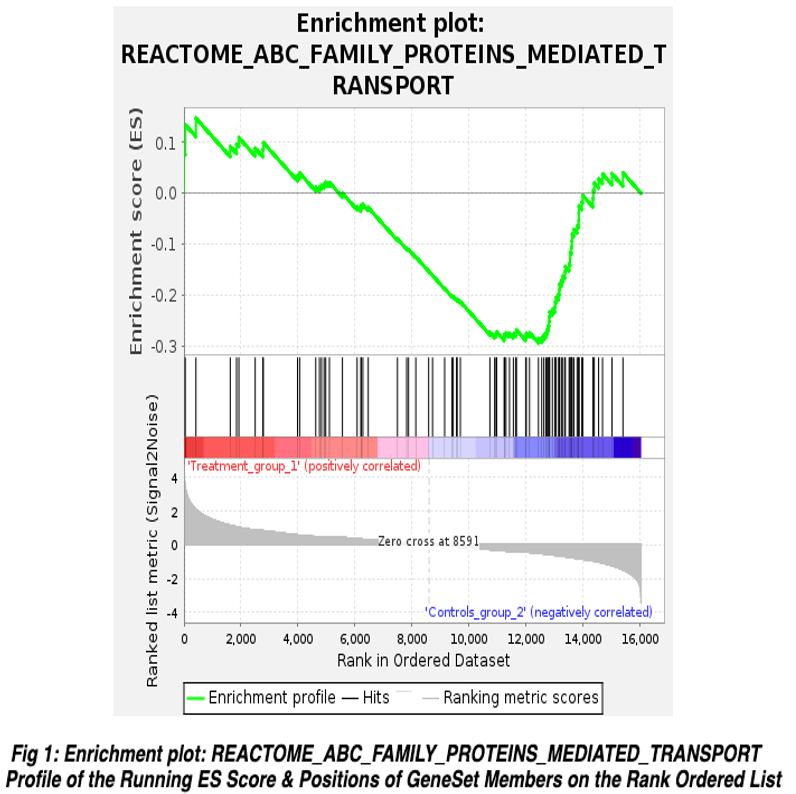
図6では、各パスウェイのエンリッチメントプロットを示します。
<pathway>.tsv
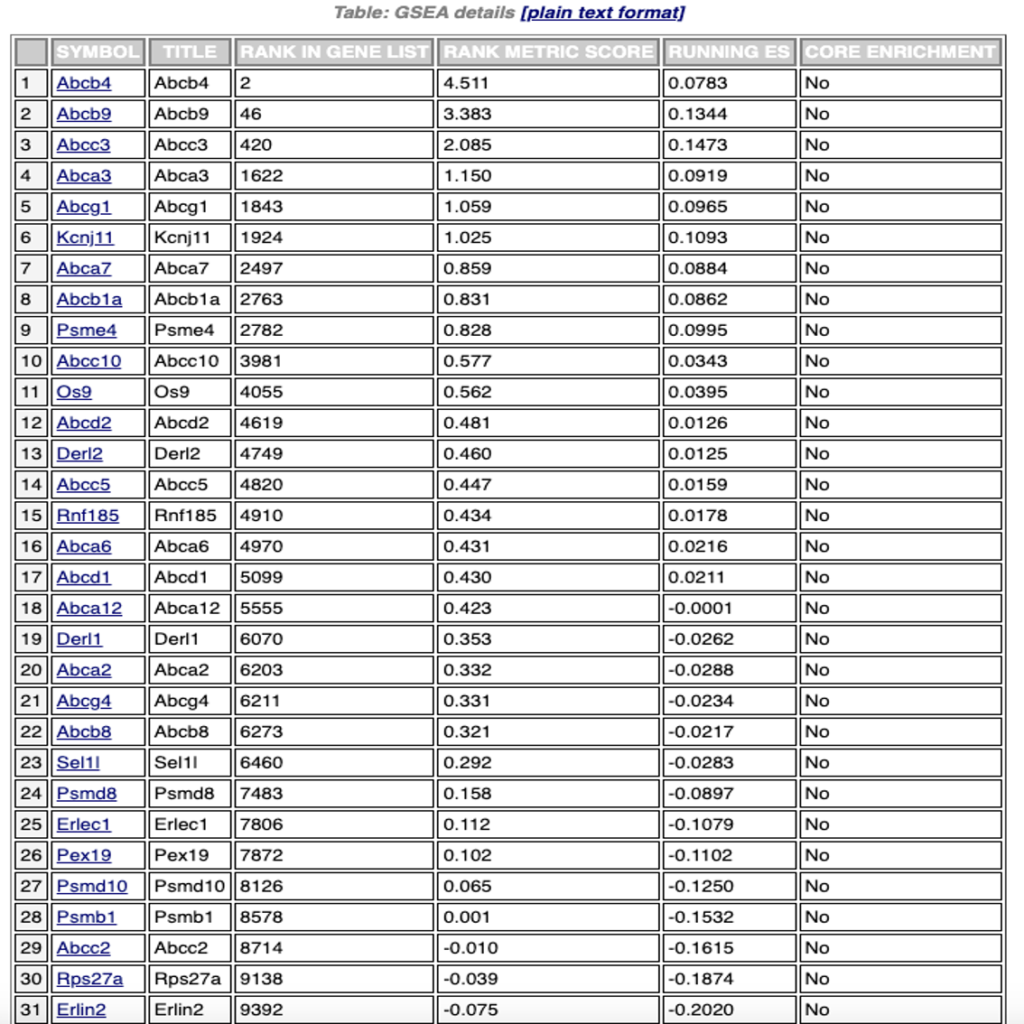
図7では、特定のパスウェイに含まれる遺伝子のリストを示します。各遺伝子がパスウェイのエンリッチメントにどのように寄与しているかを記述するメトリクスが含まれます。
REACTOME_<pathway>.png
図8では、各エンリッチメントパスウェイにおける遺伝子の発現レベルを示すヒートマップを示します。

gset_rnd_es_dist_<number>.png
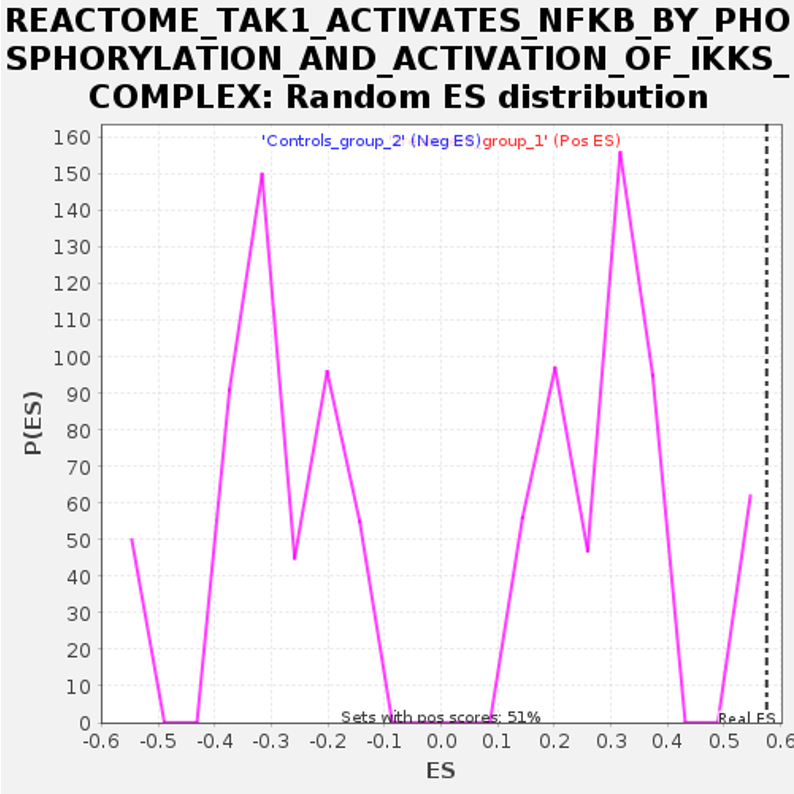
図9では、各エンリッチメントパスウェイのランダムエンリッチメントスコア分布プロットを示します。
全パスウェイの遺伝子数(gene_set_sizes.tsv)
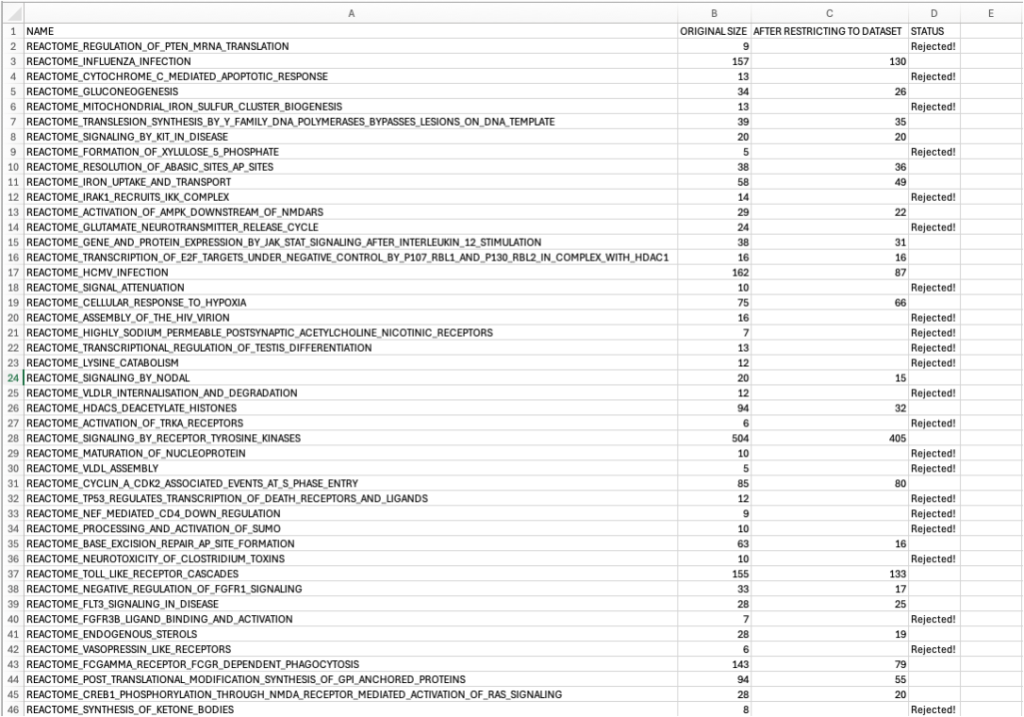
図10では、すべてのパスウェイに含まれる遺伝子数を示します。
詳細なランク付けされた遺伝子リストとメトリックスコア(ranked_gene_list_<Treatment group>_versus_<Controls group>.tsv)
図11では、ランク付けされた詳細な遺伝子リストとメトリックスコアを示します。
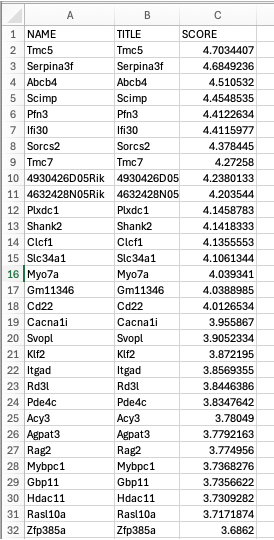
上位50遺伝子とランク付けされた遺伝子リストの相関プロファイルのヒートマップ(Heat_mal_corr_plot.html)
図12では、各表現型の上位50遺伝子のヒートマップを示します。ランク付けされた遺伝子リストの相関プロファイルも提供されます。
Heat_map_1.pngとRanked_list_corr_2.png
図12aでは、各表現型の上位50遺伝子のヒートマップを示します。また、ランク付けされた遺伝子リストの相関図も示されています(図12b)。
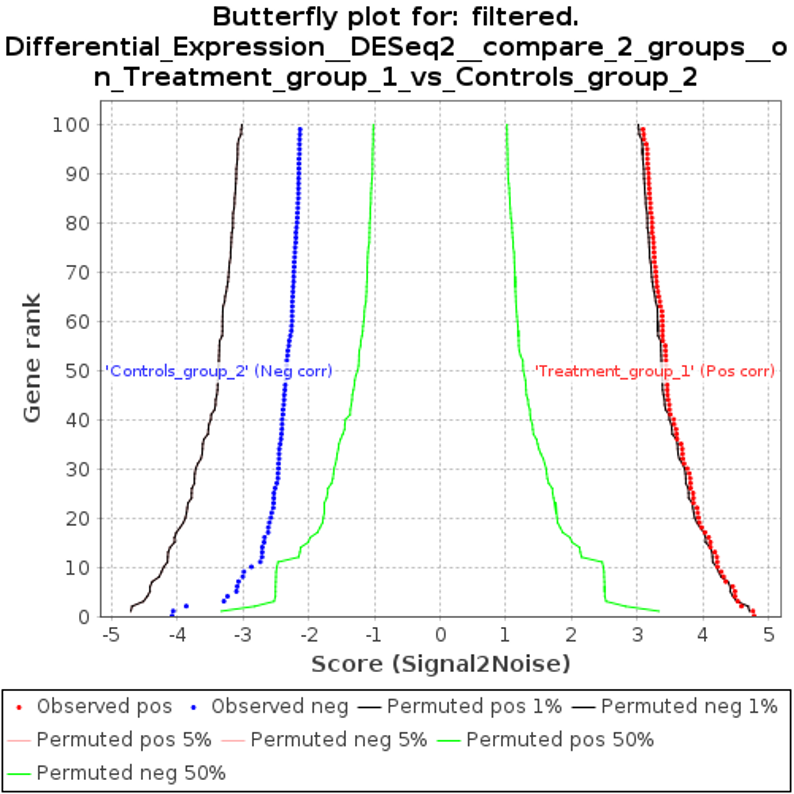
バタフライプロット(butterfly_plot.png)
図13では、有意な遺伝子がどのようにランク付けされ、異なるサンプルグループ間に分布しているかを示すバタフライプロットを示します。
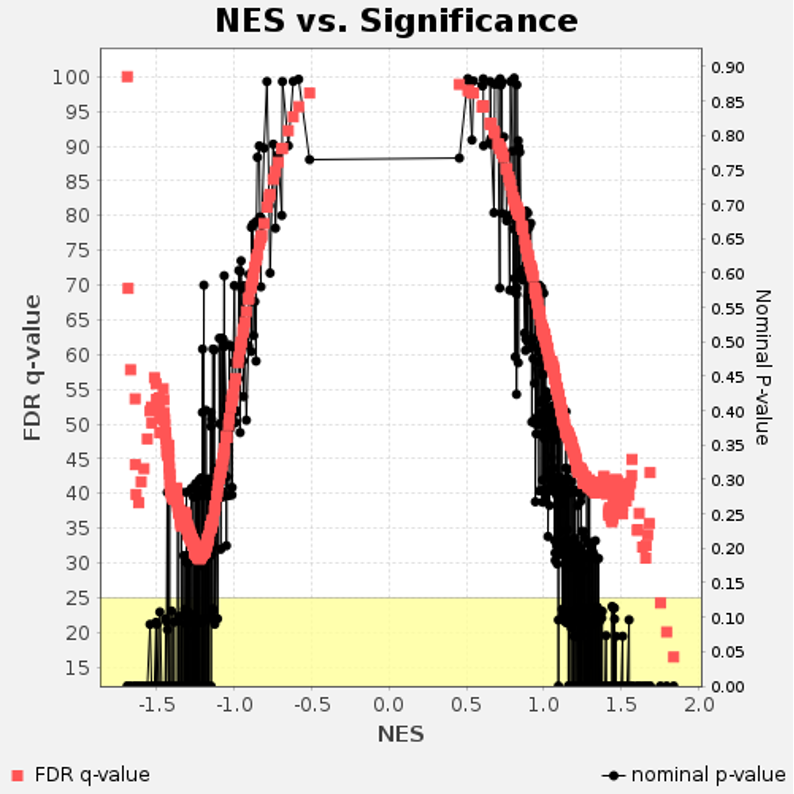
p値とNESのプロット (pvalues_vs_nes_plot.png)
図14では、NES(Normalized Enrichment Score、正規化エンリッチメントスコア)に対するp値のプロットを示します。NESはパスウェイ遺伝子セットのサイズに対して生のESを調整します。これにより異なるサイズの遺伝子セット間の比較が可能になります。
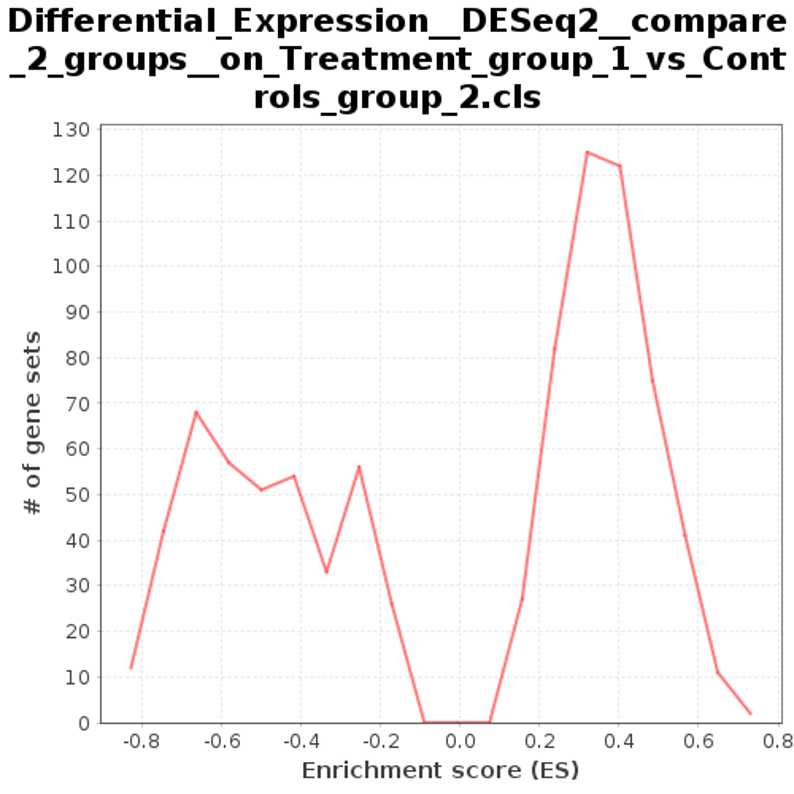
グローバルESヒストグラム (global_es_histogram.png)
図15では、パスウェイ遺伝子セット全体のエンリッチメントスコアのグローバルESヒストグラムを示します。
すべてのパラメータ設定を含むレポートファイル(GSEA_results.pathway.Gsea.rpt)
図16では、パスウェイエンリッチメント解析で使用したすべてのパラメータ設定を含むレポートを示します。

「edb」 というのサブフォルダ
このサブフォルダには以下のファイルが格納されています:
- <analysis name>.cls: クラスファイル。このファイルは、解析で比較されるサンプルグループを定義します。
- filtered.rnk: ランクリストファイル。このファイルには、異なるサンプルグループ間の発現変動によってランク付けされた遺伝子のリストが含まれています。
- gene_sets.gmt:遺伝子セットファイル。このファイルにはパスウェイエンリッチメント解析で使用した遺伝子セットが含まれます。
- results.edb:結果ファイル。このファイルにはパスウェイエンリッチメント解析の結果が格納されます(1)。
xtools.css
カスケードスタイルシートファイルです。このファイルはhtml形式のファイルの視覚的な外観を定義するために使用されますので、解析には特に関係ありません。
参考文献
1. MSigDB. (2024). Gene set enrichment analysis (GSEA) user guide. MSigDB.
関連ブログ
Basepairでパスウェイ解析(Reactome)https://basepairtech.jp/?p=2137
